こんにちは,@PKです.
今日はTukeyの多重検定をKNIMEの「R node」で処理・可視化するワークフローを紹介したいと思います.
はじめに
以前,用量反応曲線 (dose-response curve) をKNIMEの「Dose-Response (R) node」を使って処理することを紹介しました.
上記dose-response curveのフィッティングや,t検定などはKNIME上で専用のノードが用意されています.
Dose Response (R) — NodePit
Independent groups t-test — NodePit
しかし,生物学の実験では,これら以外にも様々な統計的な検定手法を使うことが頻繁にあります.
例えば,3郡以上の比較対象に対して,有意差を検出する多重検定などは.自分も実務で良く使用する検定手法です.
しかし,多重検定専用のノードはKNIMEには用意されていません…
しかし,KNIMEのR node pluginを使用することで,KNIME上で多重検定を簡単に行うことができます.
今日は多重検定の一種であるTukeyの方法をR nodeを使って紹介したいと思います.
(Rだけで完結するだろ!という意見があると思いますが,他のKNIMEのワークフローと組み合わせたい!ということで紹介しています…)
KNIME R-Project nodesのインストール
まず初めに,KNIME上でRを動かすノードをインストールしましょう.
KNIME Interactive R Statistics Integration [Feature] — NodePit
KNIMEのFile > install KNIME Extensions...を開いて,「KNIME Interactive R Statistics Integration」と検索し,インストールします.

上手くインストールができれば,以下のようにScripting > Rの下に「R projects nodes」が入ります!

R projects nodesには様々な種類があります.
それぞれのノードの意味は以下のサイトを参考にしてください.
KNIME R-Project nodes [Plugin] — NodePit
KNIME上でRを動かすときの注意点
上記RノードをインストールをしただけではKNIME上でRを動かすことはできません.以前のブログで「Dose-Response (R) node」を動かすときに紹介したように,KNIME上からRを動かす際は,R serverを使います.
以下の手順でR serverをセットアップします.
R server for knime · knime-mpicbg/knime-scripting Wiki · GitHub
あとは,File > Preferenceから,Rのパスが通っているか確認しておきます.

KNIME上でRを立ち上げる際は,Rで下記コマンドを実行しておきます.
#R library(Rserve); Rserve(args = "--vanilla --RS-enable-remote");
KNIME Workflowの紹介
それでは実際にWorkflowを紹介していきます.
とはいえ,下準備が少し大変なだけで,以下のように,非常に簡単なノード処理です.

まずは「Excel Reader」でテストファイルを読み込みます.
テストファイルは以下のように,A~Dのグループとそれぞれの値を入力しました.

それでは,「R Snippet」ノードでTukey検定を実行し,検定結果を出力します.
RでのTukey検定は以下を参考にしました.
r - Exporting TukeyHSD results - Stack Overflow
TukeyHSDはRの「multcomp」というパッケージを使うので,事前にRにインストールをしておきます.
https://cran.r-project.org/web/packages/multcomp/multcomp.pdf
「R Snippet」の設定画面で,R scriptを書いて実行します.

R script
#R snippet library(multcomp) test <- knime.in group <- knime.in$"group" value <- knime.in$"value" ANOVA_test<-aov(value~group, data=test) summary(ANOVA_test) TKHSD_test <- TukeyHSD(ANOVA_test) TK<-(TKHSD_test) TK_data<-as.data.frame(TK[1:1]) knime.out <- TK_data
KNIME独特なのは入力と出力でknime.in, knime.outを使うことくらいだと思います…
実行した後に,右クリック > view R Std outputでANOVA検定の結果を確認します.
Tukey多重検定の結果は,Data Outputで確認することができます.

無事検定ができていますね…!
続いて,「R view (Table)」でTukeyHSDの結果を可視化しましょう. ここではggplot2を使って可視化しているので,パッケージをインストールしておきます.
可視化の部分はこちらを参考にしました.
r - How to visualize pairwise comparisons with `ggplot2`? - Stack Overflow
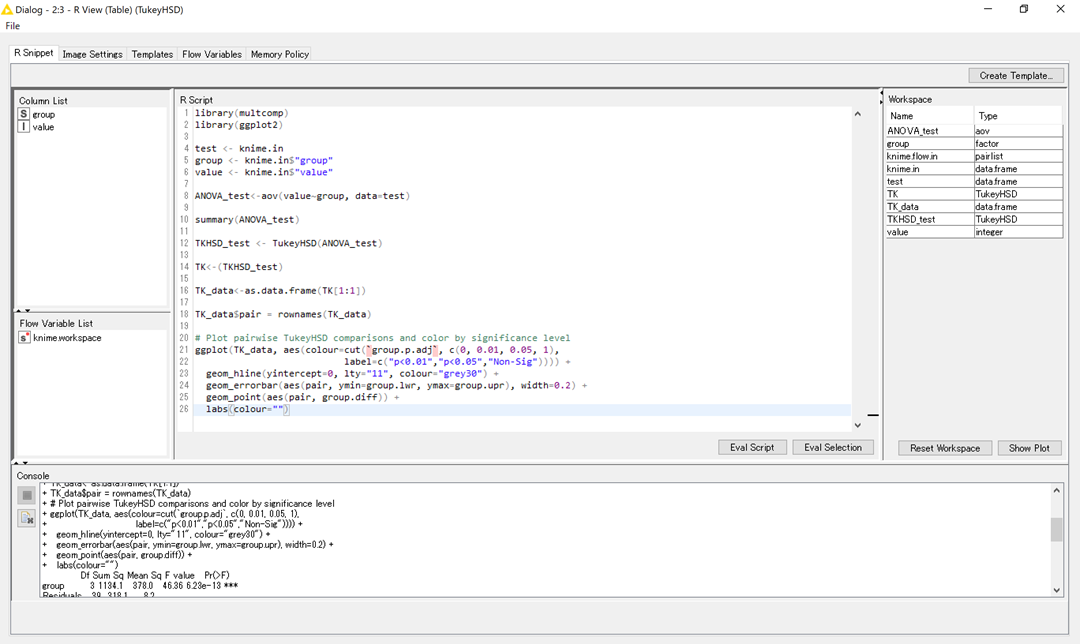
R script
# R View library(multcomp) library(ggplot2) test <- knime.in group <- knime.in$"group" value <- knime.in$"value" ANOVA_test<-aov(value~group, data=test) summary(ANOVA_test) TKHSD_test <- TukeyHSD(ANOVA_test) TK<-(TKHSD_test) TK_data<-as.data.frame(TK[1:1]) TK_data$pair = rownames(TK_data) # Plot pairwise TukeyHSD comparisons and color by significance level ggplot(TK_data, aes(colour=cut(`group.p.adj`, c(0, 0.01, 0.05, 1), label=c("p<0.01","p<0.05","Non-Sig")))) + geom_hline(yintercept=0, lty="11", colour="grey30") + geom_errorbar(aes(pair, ymin=group.lwr, ymax=group.upr), width=0.2) + geom_point(aes(pair, group.diff)) + labs(colour="")
実行した後に画像を確認してみると,Tukey多重検定の結果が図示できています!

終わりに
ここまでくると,最初からRでやろうかな…という気もしてきました.
KNIMEを使う利点は,データの前処理や,出力データの解析をプログラミング初心者でもたやすく行うことができることかな,思います.
まだまだ知らないことだらけなので,引き続きKNIMEやRの勉強していきます.