こんばんは,@PKです.
以前,KNIMEのループについて紹介しました.
t-kahi.hatenablog.com
最近,KNIMEを使って実験データの解析を自動化できるワークフローを作成しています. 出力されたデータをポチポチとEXCELに打ち込んで解析するのではなく,自動的に解析結果が出力されるので,大変便利です!
今日は,複数のデータ構造のファイルに関するループについて少し勉強になったことがあったので,紹介したいと思います.
サンプルとして以下の3つのエクセルファイルを用意しました.
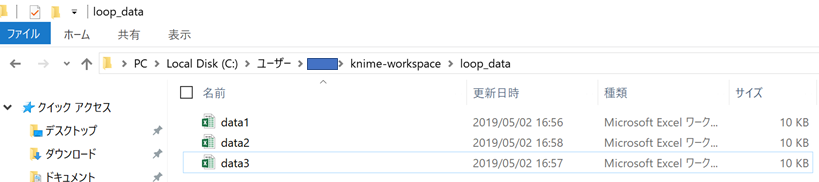
中身はこんな感じで,化合物やsiRNAのデータが含まれています.
| data1 | data2 | data3 | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| compound | conc. (uM) | data-A | data-B | siRNA | data-A | data-B | compound | conc. (uM) | data-A | data-B | ||
| A | 1 | 37 | 44 | siRNA-1 | 23 | 7 | H | 3 | 39 | 30 | ||
| B | 1 | 97 | 63 | siRNA-2 | 51 | 82 | I | 3 | 6 | 55 | ||
| C | 3 | 45 | 39 | siRNA-3 | 97 | 3 | J | 3 | 82 | 19 | ||
| D | 3 | 69 | 61 | siRNA-4 | 68 | 35 | K | 10 | 48 | 34 | ||
| E | 1 | 96 | 89 | siRNA-5 | 59 | 83 | L | 10 | 33 | 33 | ||
| F | 1 | 64 | 91 | siRNA-6 | 65 | 8 | M | 10 | 72 | 25 | ||
| G | 10 | 61 | 22 | siRNA-7 | 29 | 99 | N | 10 | 4 | 76 |
ポイントは,これらのカラムの名前(compound, siRNA, conc.(uM))や数がバラバラということです.
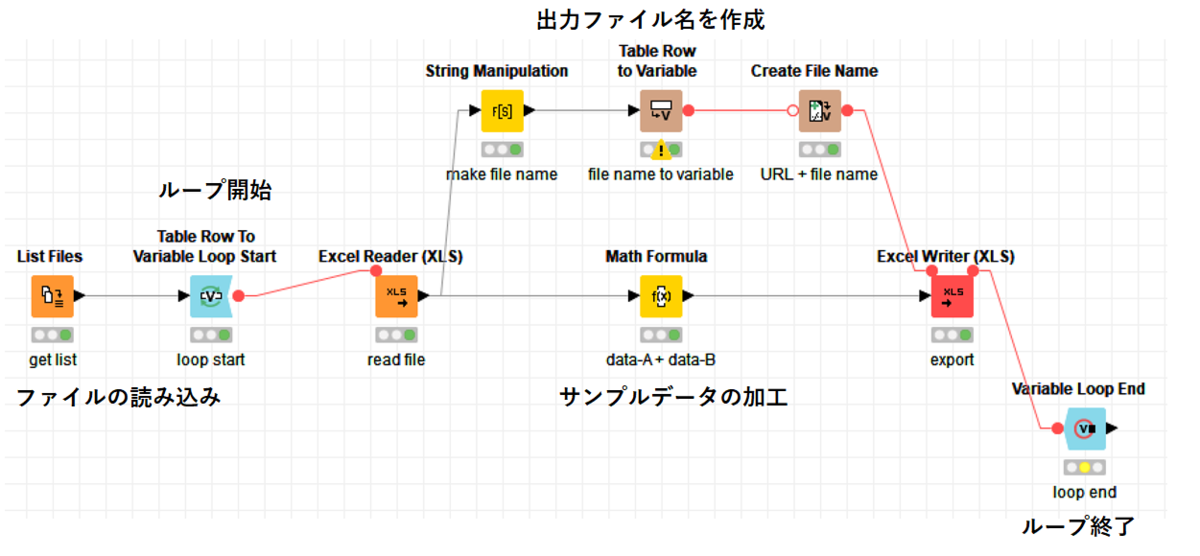
これらのデータを使って,ループ構造を含んだワークフローを作成してみます.
ループについては以前にブログで紹介しているので説明は割愛します.
以下のワークフローは,データに含まれている「data-A」と「data-B」を足し算した「data-C」を加えたデータを出力します.
ループをせずに単純にこのワークフローを出力すると,このようなファイルが出力されます. 当たり前ですね…
| compound | conc. (uM) | data-A | data-B | data-C |
|---|---|---|---|---|
| A | 1 | 4 | 62 | 66 |
| B | 1 | 77 | 68 | 145 |
| C | 3 | 36 | 7 | 43 |
| D | 3 | 30 | 85 | 115 |
| E | 1 | 40 | 91 | 131 |
| F | 1 | 24 | 35 | 59 |
| G | 10 | 78 | 29 | 107 |
これをループしてみると…loop endノードにエラーが出てしまいました
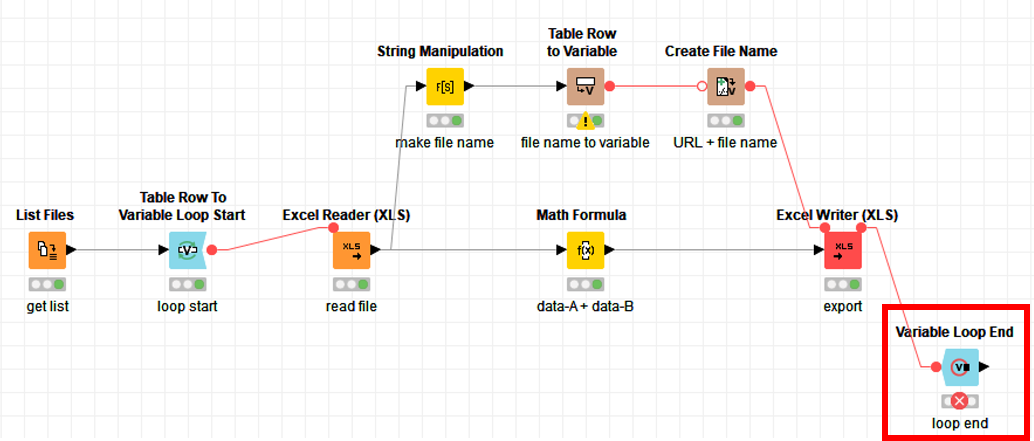
エラーメッセージを見てみると…テーブル構造が異なることによるエラーが起きていました.
ERROR Variable Loop End 2:13 Execute failed: Output table's structure differs from reference (first iteration) table: different column counts 11 vs. 12. Have the input variables changed in number, name or type?
variable loopはテーブル構造が同じものしかループすることができないのです…!
今回は,compound,siRNAなど異なるデータ構造を持ったファイルがこれらのエラーの原因のようです.
しかし,実際の実験データは様々なテーブル構造を含んでおり,それらをまとめて自動化したいですよね.
解決策を探していると同じようなエラーの人が何人かいるようで,,, X-Aggregator Execute failed: Input table's structure differs from reference (first iteration) table: different column counts - KNIME Analytics Platform - KNIME Community Forum
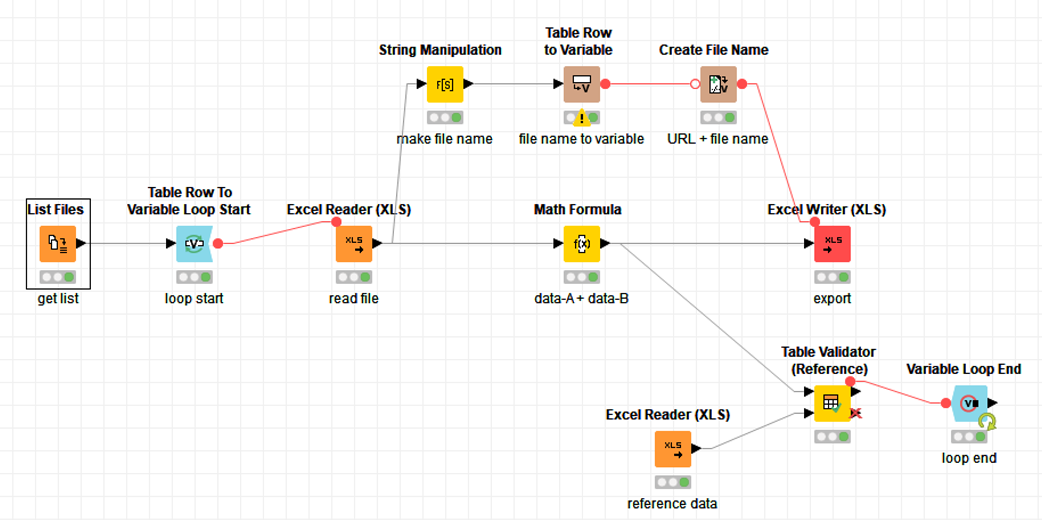
これらのフォーラムを参考にしながら,異なるテーブル構造でもループができるようにしてみたのが以下のワークフローです.
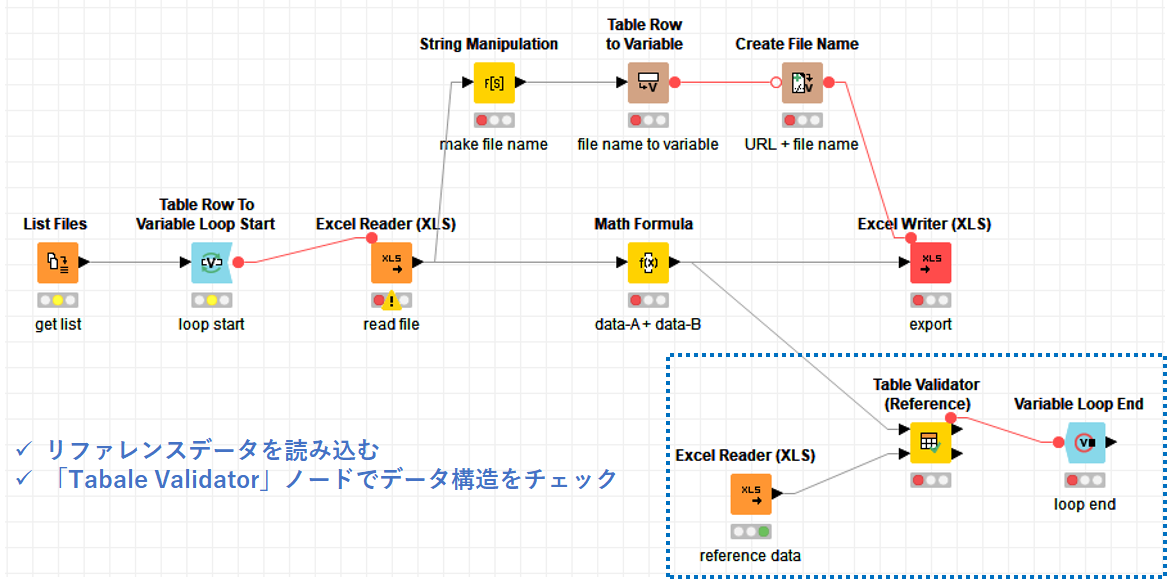
ポイントは青い点線部分のノードの部分です. 「Tabale Validator」ノードを使って,テーブル構造を一定に保つことができます.
端的に言うと,「データ構造が異なるのが問題なので,ループ終了時にダミーテーブルを使ってデータ構造を一定に保つ」ということをしています.
Tabale Varidatorノードは,テーブル構造やデータの内容を保証することができるノードです.
Table Validator (Reference) — NodePit
今回は「Tabale Validator(reference)」ノードを使用しているのでリファレンスデータをベースとして,入力ファイルのデータ構造をバリデーションします.
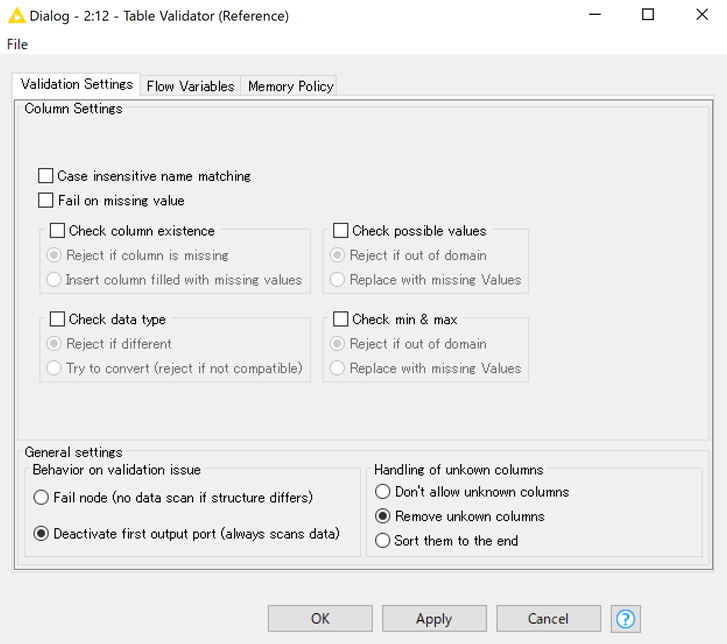
「Tabale Validator(reference)」ノード設定を開くといくつか設定する項目があります.
今回は入力データがリファレンスデータが異なっている場合に,修正できればよいので,以下のように「handling of unknown column」⇒「Remove unknown column」に設定しておきます.
この設定で解析をするとエラー無くデータが出力することができました!
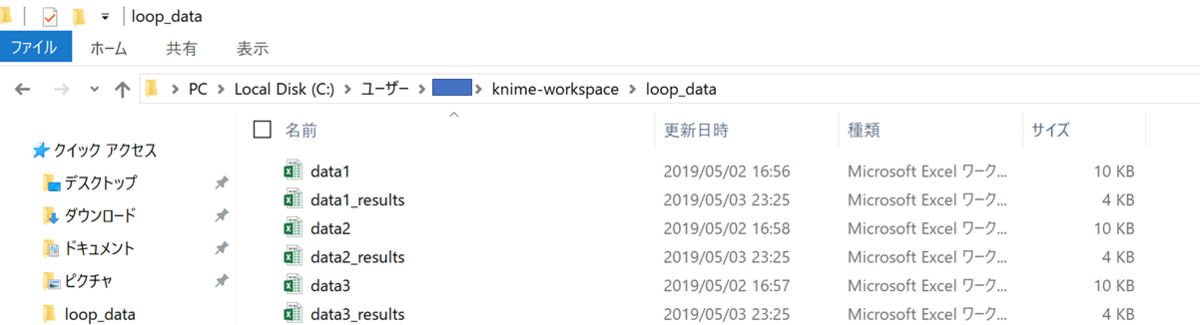
これでデータ構造が異なるファイルでもループを回すことができます.
実際に自分の実験データもこのtable varidatorを使って実験データの解析をしています.
ルーチンの評価ではなく,初期検討など,毎回データの種類が変わるような実験をするような場合には有効かもしれません.