こんばんは,@PKです.
今回はハイコンテントアナリシスで撮影した画像に含まれているノイズやぼやけた画像を検出する手法について紹介したいと思います.
はじめに
上記記事で各化合物処理による細胞小器官(=オブジェクト)の特徴量の変化量(Z-score)を取得しました.
また,得られたデータはHeatmapやImageViewerで可視化することを紹介しました.
このHeatmapで得られたデータを見ていると,全体的に特徴量が大きく変動する化合物があることがわかりました.
Heatmapの一部を左図で示していますが,例えば,LY-83583とNoscapineという薬剤処理群は多くの特徴量の値が変動しています.
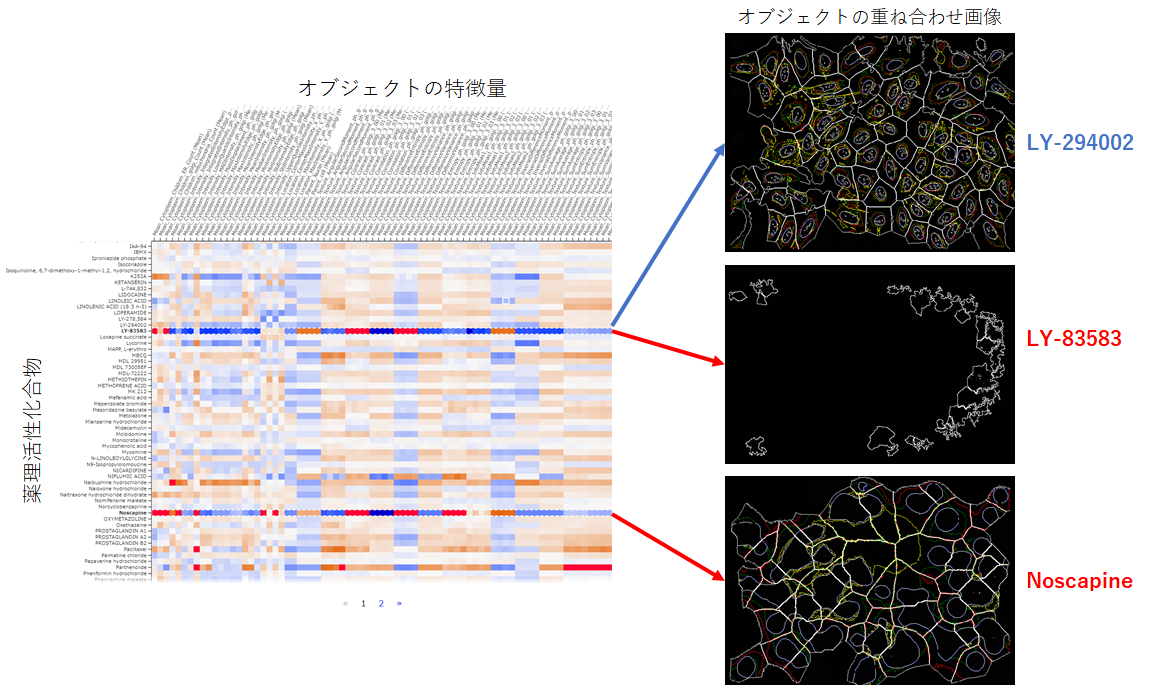
ImageViewerで化合物処理のオブジェクト重ね合わせ画像を確認してみると,比較対象のLY-294002とは異なり,LY-83583とNoscapineは,細胞の認識のされかたが大きく異なっています.
そこで,それぞれのヘキスト染色のrow dataを比較してみたのが以下の図です.

LY-83583とNoscapineはどちらもフォーカスがうまく合っていないことによる画像取得の異常が起きていることがわかりました.
特にHCAのように大規模で画像を解析する場合はこのような画像のピントが合っていないことによる擬陽性・偽陰性が良く生じます.
もう一つの例として以下の図をお示しします.
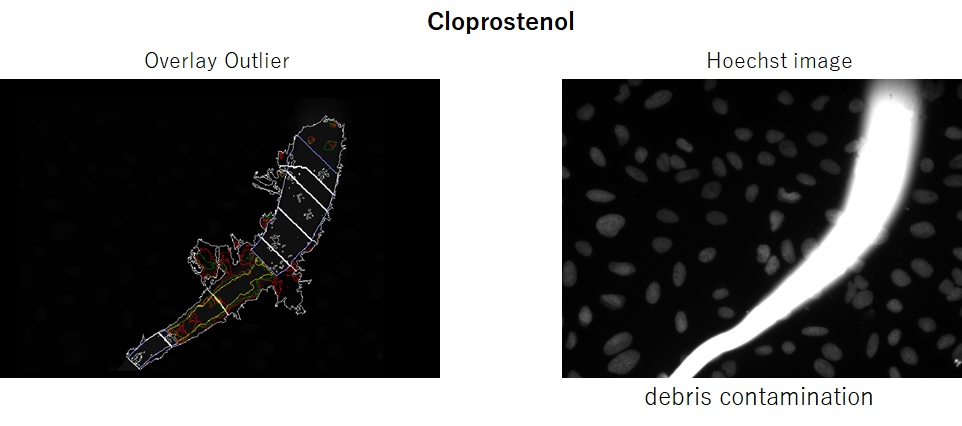
こちらはゴミが混じっていたために検出がうまくいかない例です. こちらも画像染色でよくある例です.
このような画像から解析した結果を基にデータでは,解析結果に変なバイアスを与えてしまう可能性があるため,どの画像データに異常があるかを早めに知っておく必要があります.
そこで,ゴミやピンボケの画像を検出できるモジュールとして「MeasureImageQuality」がCellProfilerに用意されています.
CellProfilerの「MeasureImageQuality」モジュールについて
「MeasureImageQuality」モジュールは画像の質に関する様々な情報を取得することができます.
Measurement — CellProfiler 3.0.0 documentation
MeasureImageQualityモジュールを以下に示します.
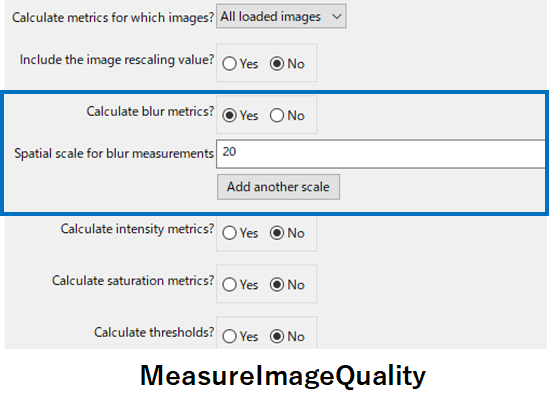
このモジュールで画像のぼやけに関する部分を青枠で囲みました.
「Calculate metrics for which images?」 では,どの画像を解析するのか選択します.「All loaded images」を選ぶと読み込んだ画像全てを解析します.
続いて「Calculate blur metrics?」でBlur(ぼかし)に関する解析を行うかを選択します.
「Spatial scale for blur measurements」の項では計算したいピクセル数を設定します.
例えば20という値を設定した場合は,「LocalFocusScore(後で説明します)」という数値は20×20 pixel内で測定されます.
また,「Correlation」は20 pixel離れたpixelに関して相関度を測定します.
このピクセル数を高くすることで大きなパターンのぼやけを測定し,小さな値は局所的なぼやけを測定することができます.
一般的には,対象とするオブジェクトと同じくらいのサイズで設定するのが望ましいそうです.
この値は複数設定することができるのでいくつか値を比べることもできます.
それでは先ほど例に挙げた画像をCellProfilerの「MeasureImageQuality」モジュールで解析してみます.
「Spatial scale for blur measurements」の値はとりあえず20にしました.
その結果を以下に示します.

- 「DNA focus score @20」:画像全体のピクセル強度の正規化分散
⇒画像全体の強度分散を平均強度で割ったもの
⇒値が高いほど,ぼやけ度が低い - 「DNA local focus score @20」:画像のサブ領域ごとに算出したピクセル強度の正規化分散を平均化した値
⇒異なる視野の画像間の比較でより有効 - 「DNA correlation @20」:画像のサブ領域ごとの空間強度分布の相関値
⇒画像がぼけていると、隣接する画素間のより相関するのでcorrelation 値が高くなる - 「PowerLogLogSlope」:画像の対数パワースペクトルの傾き
⇒パワースペクトルは画像の周波数情報を含んでおり,スロープは画像のぼやけ度を表す
⇒傾きが大きいほど低い周波数情報を含み,ぼやけ度が高い
上記の結果を見てみると…それぞれ上記の測定値はフォーカスがあっている画像と比べるとぼけている画像を認識できています.
今回例として挙げた3つの画像を正しく分類できている測定値としては「DNA correlation @20」がよさそうです.
KNIMEを使ってぼやけた画像をデータセットから除去する
せっかくなので「MeasureImageQuality」の解析結果をKNIMEで処理するワークフローを作成してみました.
データは以前紹介したデータセットの一部(20585w1~W5:全3456枚)を使用しました.
【CellProfiler】薬理活性化合物を処理した細胞染色画像から特徴量を抽出する① - t_kahi’s blog BBBC022: Human U2OS cells – compound-profiling Cell Painting experiment
CellProfilerのパイプラインは以下のようにシンプルに「MeasureImageQuality」だけを測定しました.
また,Spatial Scaleは10,20,40の3点おきました.
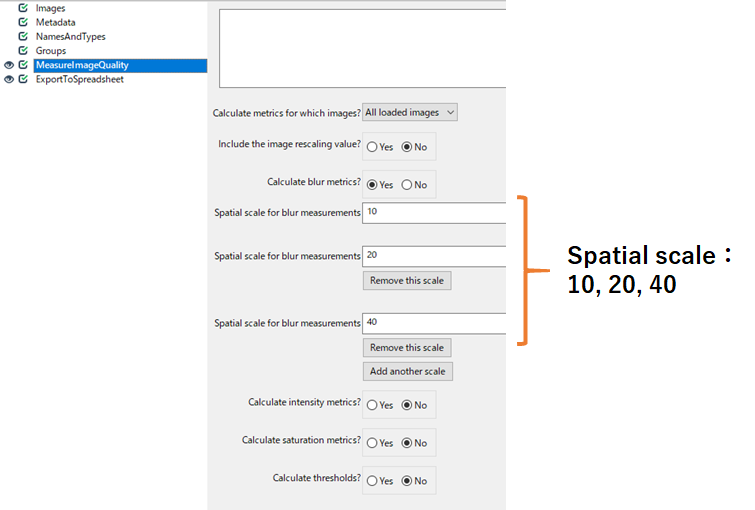
自分のPCでだいたい4時間くらいで解析が終わりました.
この解析結果を以下のワークフローで処理していきます.
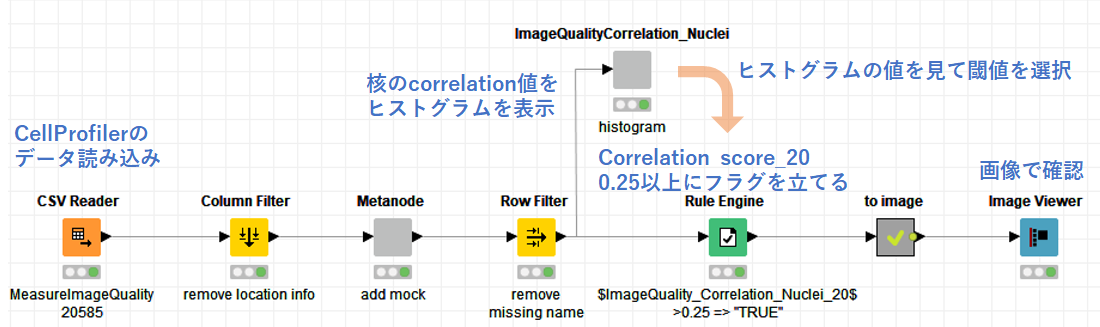
上図で青字で示したように,
- 画像を読み込み,データの前処理をする
- 核のcorrelation scoreをヒストグラム表示
- ヒストグラムから閾値を決める
- correlation score > 0.25にフラグを立てる
- フラグを立てた画像をImage viewerで表示して確認
という流れで進めていきました.
Correlation score値のヒストグラムについて
2.ではNuclei Correlation_10, 20, 40のヒストグラムをそれぞれ示しています.
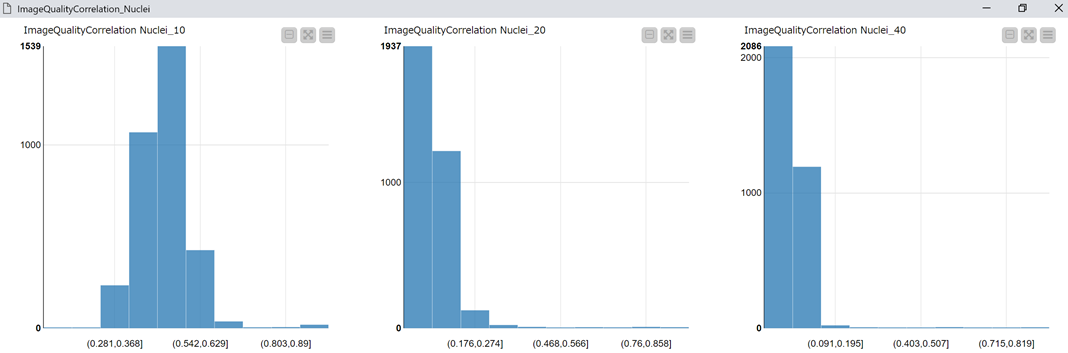
核の直径は20~40程度なので,やはりSpatial scale値は20, 40のほうが精度がよさそうです.
今回はspatial space=20の場合のヒストグラムに着目します(以下図).
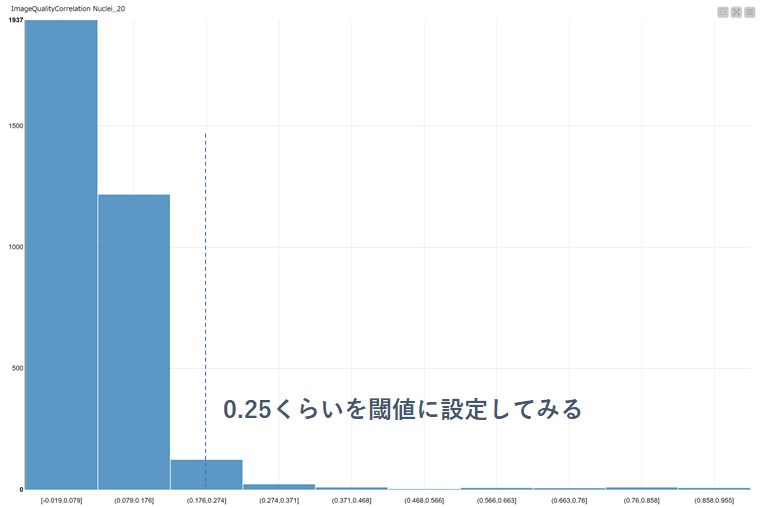
ぼやけた画像はそこまで多くはないので,0.25くらいを閾値として設定してみました.
個の閾値を「Rule Engine」ノードでNuclei Correlation_20が0.25以上の画像にフラグを立てます.
$ImageQuality_Correlation_Nuclei_20$>0.25 => "TRUE"
ImageViewerで画像を確認
ここで選択した画像を最後にImageViewerで確認します.
結構ゴミが入っていたり,ぼやけた画像,ノイズの画像を検出できていることがわかりますね!!
そこまでぼやけてないような画像も含まれていそうなので,そのあたりをどう分けるかが課題でしょうか…
最後に
実験データのQCは,アッセイを行う上で非常に重要なステップです.
HCSも同じで質の良い画像データをきちんと選ぶことは,その後の解析結果の信頼性を高めると思います.
今回MeasureImageQualityを勉強したので,今後も活用して質の高いHCSができればいいなあと思っています.