こんばんは,@PKです.
今日はKNIMEでKolmogorov–Smirnov検定(KS-test)を行うWorkflowを紹介したいと思います.
Kolmogorov–Smirnov検定(KS-test)とは
Kolmogorov–Smirnov検定とは,2つの標本の分布が一致しているかどうかを検出する検定手法です.
2つの標本についてそれぞれの母集団の確率分布が一致しているかどうか、またはある標本の母集団の確率分布が帰無仮説で提示した確率分布と一致しているかどうかを検定すること コルモゴロフ=スミルノフ検定 | 統計用語集 | 統計WEB
あまり馴染みが無い方もいるかもしれませんが,実はKS-testはHigh Content Analysisの分野でも良く使用されています.
https://journals.sagepub.com/doi/full/10.1177/1087057114526432/
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4103836/
KNIMEではこのKS-testを行うためのノードがあります.
nodepit.com
Workflowの概要ではこのノードの使い方や前処理のやり方等をご紹介します.
KNIMEworkflow概要
ご紹介するKNIME Workflowの概要です.

まず「Table Creator」でサンプルデータを読み込みます. その後,データの全体を把握するために,累積度数分布図を「R View」で表示します.
その後,データをループ処理を行いながら「Kolmogorov–Smirnov Test」を行います.
左下図がサンプルデータ,右下図が累積度数分布図を示しています.
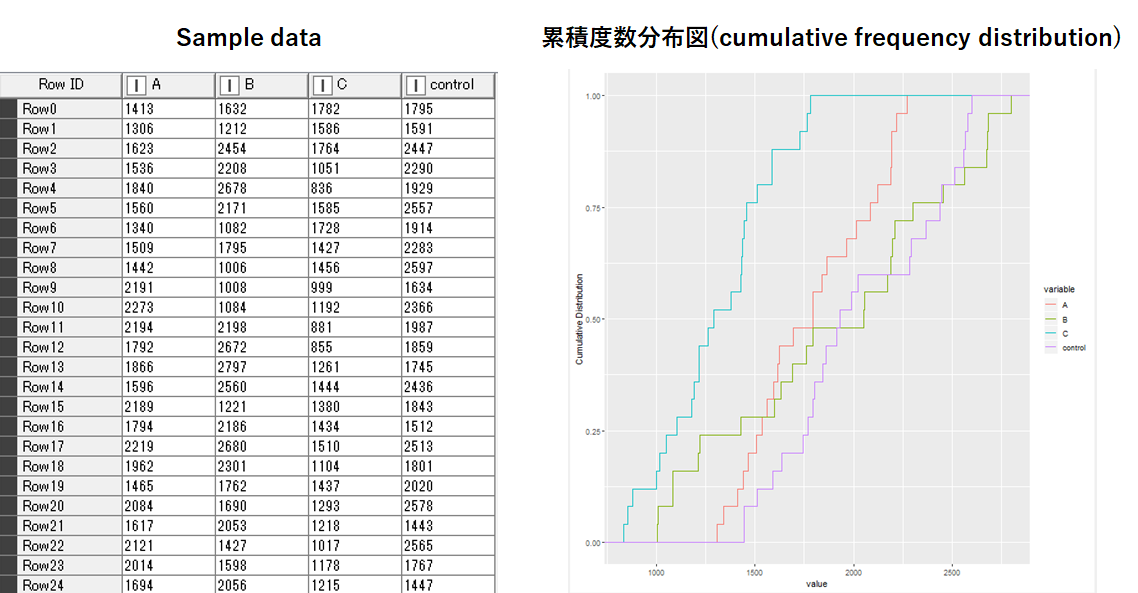
サンプルデータは,コントロールと3つの被験物質(A~C)のそれぞれの測定値を示しています.
これら測定値を表で眺めるだけでは差がわかりずらいので,累積度数分布表示で表します.
#R View library(ggplot2) library(reshape2) data <-data.frame(knime.in) data.m <-melt(data) #カラムをまとめる ggplot(data.m, aes(value, color = variable))+ stat_ecdf(geom = "step") + labs(x="value",y="Cumulative Distribution")
累積度数分布表示のイメージがわかない方は以下を参考にしてください.
統計学入門−第2章
累積度数分布図の結果から,controlとA, Cは差がありそう.controlとBは同じような度数分布をしていることがわかります.
これをKS-testで実際に検定してみます.

今回は,controlとA,controlとB,controlとC,というようにそれぞれKS-testを行います.
そのため,まずは検定を行うカラムを抜き出すことを「Column List Loop Start」で行います.

ループしたいカラム(A,B,C)を選択し実行すると,上右図のように1回目のループ処理ではcontrolとA列が選択されます.
続いて,「Column Rename (Regex)」ノードでターゲットのカラムを”ANONYMOUS_COLUMN”に変更します.

「Column Rename (Regex)」はループ処理時に便利です.
以下記事も参考にして下さい.
【KNIME】Transpose rows to multiple columns by category - t_kahi’s blog
いよいよ,「Kolmogorov–Smirnov Test」ノードを使用します.
設定は非常に簡単で,優位水準を決め(default : 0.05),測定する値を含んだカラムを指定します(左下図).
ここで,カラム名を”ANONYMOUS_COLUMN”にすることで,ループ処理の際にエラーが出ることなく進めることができます.

実行すると,右図のように,帰無仮説の棄却結果,統計検定量(累積確率の差の絶対値の最大値),p値が得られます.
controlとAのKS-testの結果はp値が0.05以下ということで,帰無仮説が棄却され,2つの分布は異なるという結果になりました.
結果をこのまま出力するのが難しいので,少し加工します.
「Transpose」で行列を入れ替えて,「Column Rename (Regex)」で再びカラム名を”ANONYMOUS_COLUMN”→”A"に戻します.

「Loop End (Column Append)」で上記処理を繰り返した結果,左下図のようになりました.
累積度数分布の結果(右下図)とも一致していますね.

まとめ(とおまけ)
KNIMEでKolmogorov–Smirnov検定を行う Workflowを紹介しました.
非常にシンプルな統計手法なので,ノードを使って処理するのはやりやすいと感じました.
ちなみに,Kolmogorov–Smirnov検定はRにも用意されており,「R snippet」で非常に簡単に同じことを行うことができます.
https://stat.ethz.ch/R-manual/R-patched/library/stats/html/ks.test.html
#R snippet data <- data.frame(knime.in) ks_test <- ks.test(data$control,data$ANONYMOUS_COLUMN) knime.out <- data.frame(ks_test["statistic"],ks_test["p.value"],ks_test["alternative"])