こんばんは,@PKです.
前回の記事では線形判別分析(LDA)のKNIME Workflowをご紹介しました.
www.t-kahi.com
LDAは複数のクラスのデータを最も良く判別できるように線を引く,というイメージでした.
異なる判別分析の手法として決定木(decision tree)による分類があります.
今回は決定木を使って判別分析を行うKNIME Workflowをご紹介します.
決定木 (decision tree) とは
決定木 (decision tree)はツリーモデル (tree-based model) の一種で,データの説明変数を条件によって分類し,木のようにデータを分類していく手法です.
決定木は判別・分類問題での呼び方で,回帰分析の場合は回帰木 (regression tree)と呼ばれています.
決定木の詳細については下記記事を参考にして勉強しました.
データ解析・マイニングとR言語
R超入門 - Rのインストールから決定木とランダムフォレストによる分析まで - Qiita
機械学習③ 決定木 (Decision Tree)のまとめ - Qiita
今更感あるけど決定木について調べたのでまとめる - St_Hakky’s blog
KNIMEのDecision Tree nodeの紹介動画が相当クオリティ高いのでこちらを見れば十分理解できるかもしれません…
Decision Tree | KNIME
それではKNIME Workflowで決定木の例を見ながら細かい部分をご紹介していきます.
KNIME Workflowの概要
以下に今回のKNIME Workflowをお示しします.
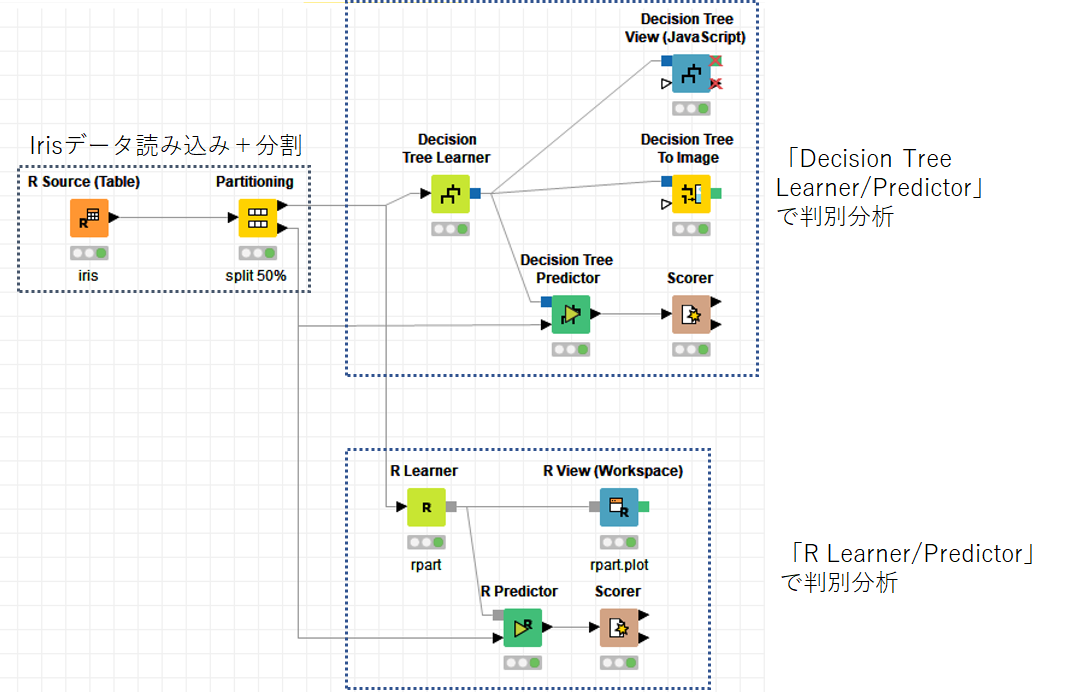
今回もKNIMEの決定木用ノードとRノードそれぞれのWorkflowを紹介します.
前回と同様にirisデータ読み込み,半分に分割した後に,「Decision Tree Learner/Predictor」で判別分析を行うWorkflowと,「R Learner/Predictor」で判別分析を行うWorkflowを紹介します.
irisデータの読み込みはいつも通りです.
#R Table knime.out <- iris head(iris)
> knime.out <- iris + head(iris) Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 2 4.9 3.0 1.4 0.2 setosa 3 4.7 3.2 1.3 0.2 setosa 4 4.6 3.1 1.5 0.2 setosa 5 5.0 3.6 1.4 0.2 setosa 6 5.4 3.9 1.7 0.4 setosa
実際のWorkflowの説明に移ります.
「Decision Tree Learner/Predictor」で判別分析
「Decision Tree Learner/Predictor」を使ったWorkflowを以下に示します.
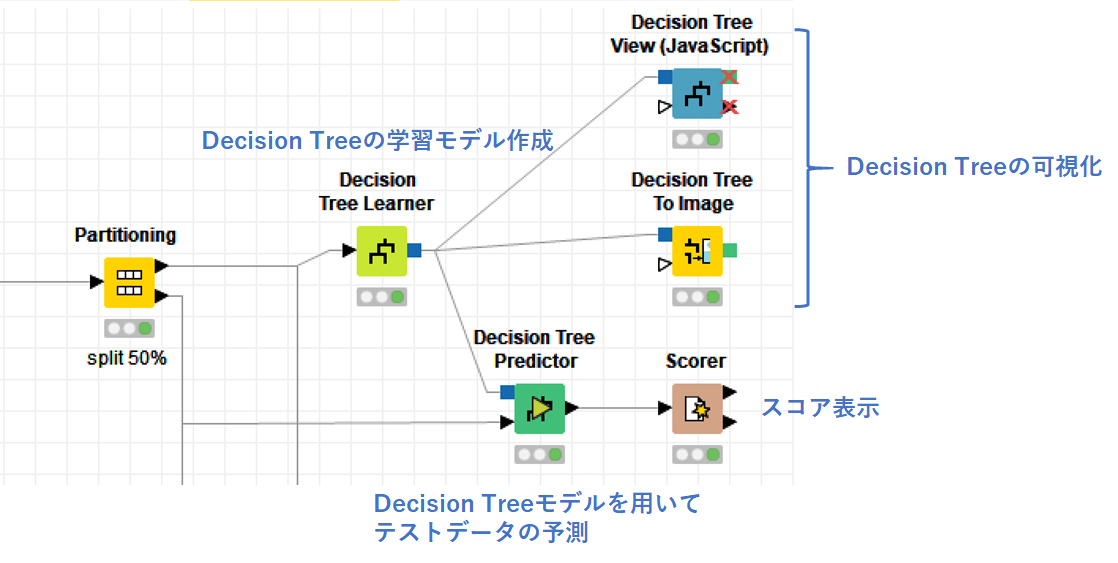
まず,「Decision Tree Learner」で学習データを読み込んでモデルを作ります.
nodepit.com
設定画面と,主要な設定部分を以下に示します.
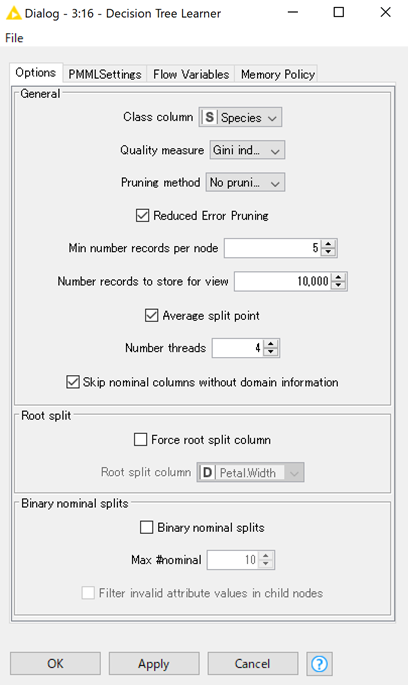
- Class column:分類するクラスのカラムを設定する
- Quality measure:データ分割の手法( "Gini Index" or "Gain Ratio")
- Pruning method:剪定の有無( "Minimal Description Length" (MDL) か無しを選択)
- Reduced Error Pruning:単純な剪定手法が適応される(デフォルト☑)
- Min number records per node:ノードにおける最小数の個体数
- Average split point:数値データの分割時に平均値を使う(デフォルト☑)チェックが無い場合は小さい数値カテゴリー内の最大値が選ばれる
- Force root split column:どの列で分割するのが早いかわかっている場合はチェックをつける(特にわからなければチェックを付けない)
- Binary nominal splits:名義(nominal)尺度の変数を2分岐するか
Class columnには分類をしたい"Species"を選択します.
Quality measureの"Gini Index"と"Gain Ratio"はデータ分割の主な手法です.
後ほど紹介するRのrpartパッケージでは"Gini Index"を用いた CART (classification and regression trees)アルゴリズムを使用しています.
[決定木]ジニ不純度と戯れる - Qiita
こちらのほうが一般的とのことなので,ここでも"Gini Index"を使用します.
続いて"Pruning method/Reduced Error Pruning"では剪定(Pruning)について設定します.剪定は無駄なデータ分割や過学習を避ける意味で使用されます.
また,重要な設定として"Min number records per node"があります.
こちらは分岐先の個体数を設定できます.
今回は5に設定してあるので,個体数が5以下になったらこれ以上分割が起きなくなります.過学習を防ぐためにデータ数に応じて設定する必要があるようです.
設定後に実行するとモデルが作成されます.
学習データがどのように分類されたかを確認するには,以下の可視化ツールを使用します.
nodepit.com
nodepit.com
「Decision Tree To Image」で作成した分類木を以下に表示します.

"Setosa"はきれいに分けられています.
"Versicolor"と"Virginica"は少し誤判別していますが,おおむね分けれられているようです.
「Decision Tree View」のinteractive viewsの結果は最後に動画で紹介します.
作成したモデルを使って,「Partitioning」で分けた半分のテストデータの予測を「Decision Tree Predictro」で行います.
「Decision Tree Predictro」の設定画面と実行結果を以下に示します.
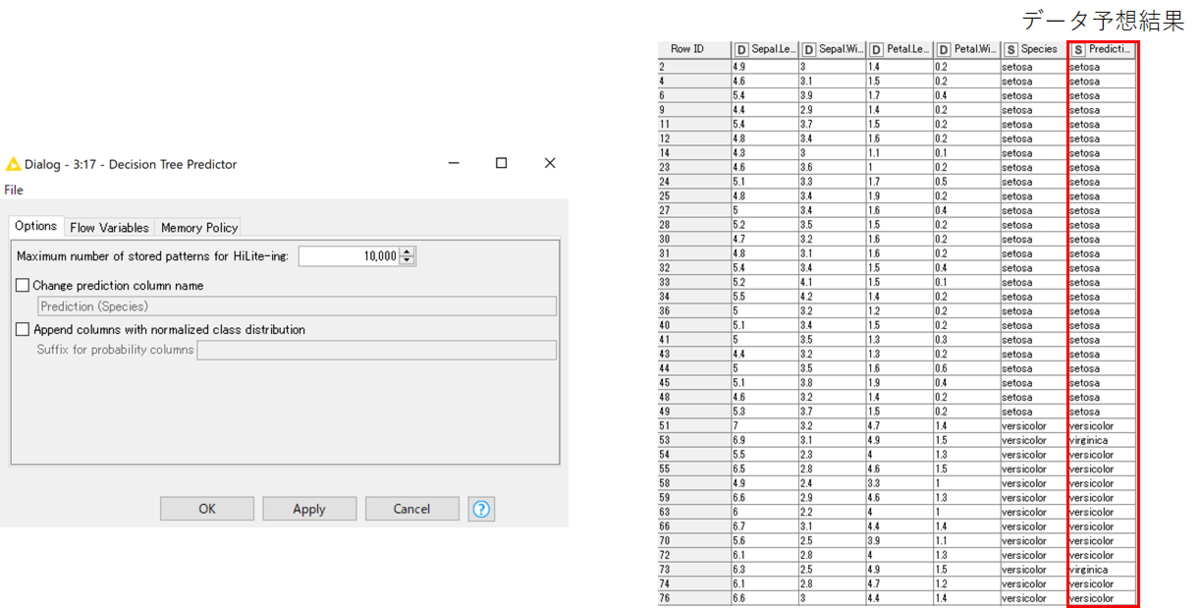
判別分析の予測結果が最後のカラムに追加されました.
どの程度判別分析がうまくいっているかを確認するために,「Scorer」を使用します.
以下に設定画面(左図)と実行結果(下図,右図)を示します.
nodepit.com
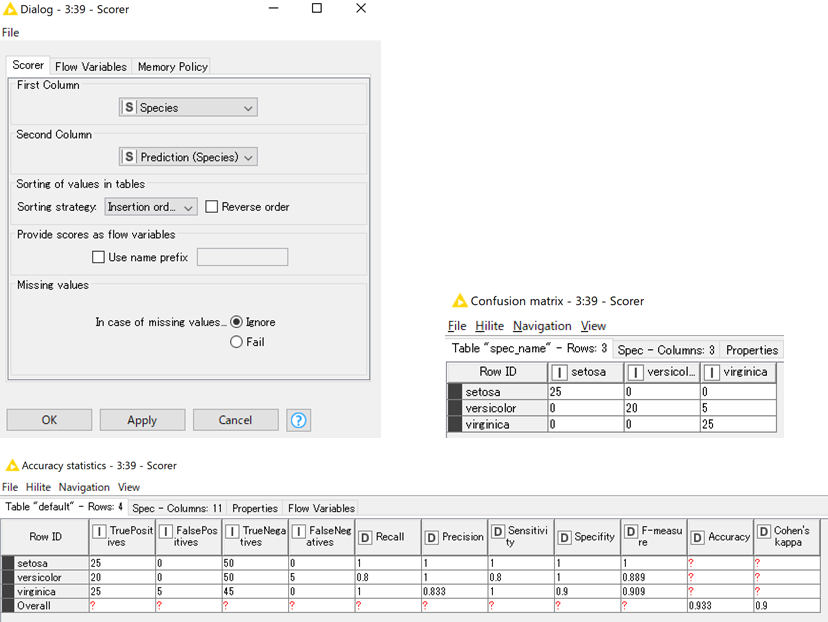
「Scorer」で比較したい2つのカラムを選択し実行すると,実行結果が返されます.
「R Learner/Predictor」のrpart関数を使って判別分析
続いて,同じ決定木による判別分析を「R Learner/Predictor」で行います.
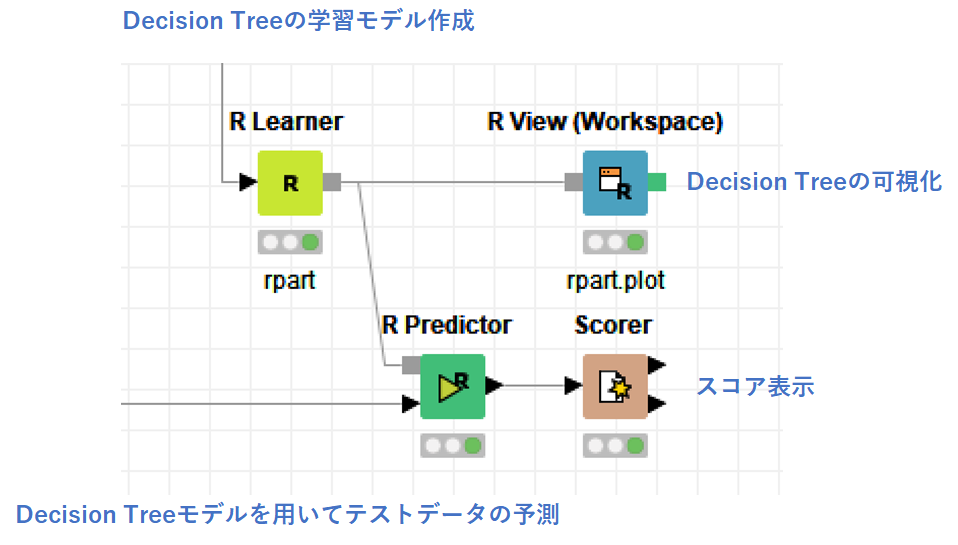
使用するパッケージとして一般的に使用されているrpart(recursive partitioning and regression trees)を選びました.
rpart function | R Documentation
まず「R Learner」で決定木モデルを作成します.
# R Learner library(rpart) data <- knime.in iris.rp<-rpart(data$"Species"~.,data[1:(ncol(data) - 1)],minsplit=5,cp=0) printcp(iris.rp)#上記#交差確認法の結果を表示 knime.model <- rpart(data$"Species" ~ ., data[1:(ncol(data) - 1)])
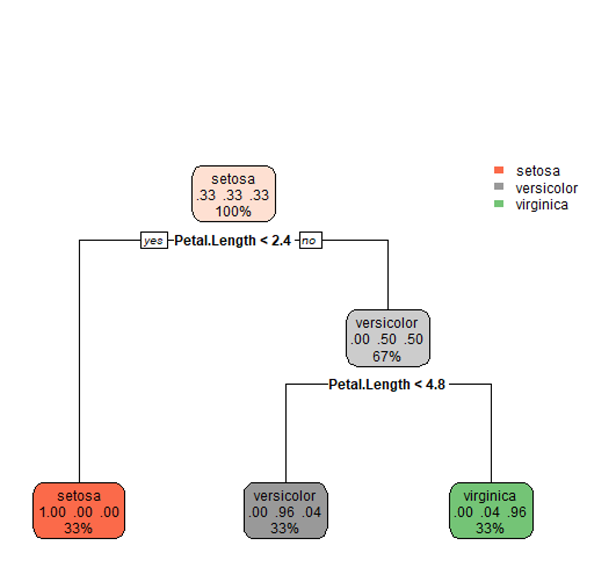
作成した分類木を「R View」で表示します.
# R VIew library(rpart.plot) model <- knime.model rpart.plot(model)
続いて「R Predictor」でテストデータを分析します.
テストデータの分析は"predict"を使用するだけなので非常に簡単です.
# R Predictor knime.out <- cbind(knime.in, predict(knime.model, knime.in,type="class"))
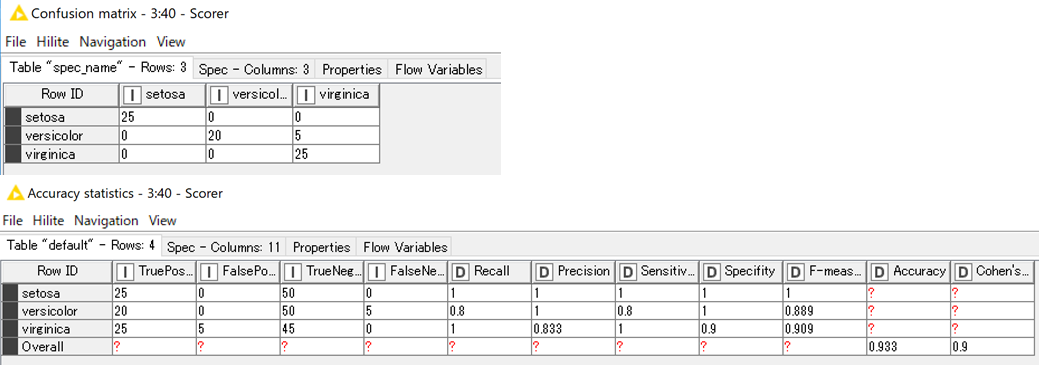
最後に先ほどと同様に「Scorer」でテストデータ解析の結果を確認し.同じ結果が得られたことがわかりました.
まとめ
KNIME Workflowを使えば決定木の判別分析をかなり簡便に行えます.
KNIMEノードだけで学習するよりも,RやPythonを使った学習も一緒に学んでおけばいろいろと応用が利くような気がします.
最後にWorkflow全体の動画を示します.
youtu.be