こんばんは,@PKです.
前回の記事では,Support Vector Machine (SVM) のKNIME Workflowについて紹介しました.
www.t-kahi.com
今回はSVMのKNIME WorkflowをRやPythonのノードを使って行うWorkflowを紹介します.
KNIME Workflow概要
Workflowの概要を以下に示します.
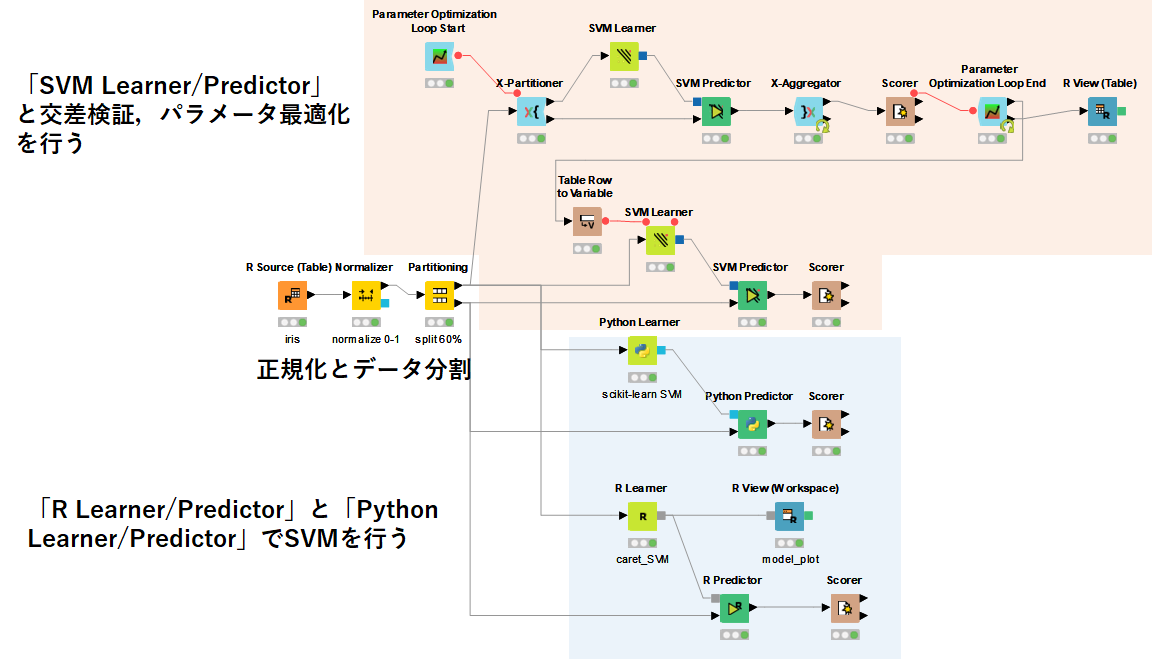
前編では「SVM Learner/Predictor」を使って,交差検証やパラメータ最適化,SVMのモデル構築を行って,実際のテストデータを解析する手法を紹介しました.
後編では,RとPythonをそれぞれ使ってKNIME上でSVMを実行するWorkflowを紹介していきます.
Rはcaretパッケージを,Pythonはscikit-learnライブラリを用いてSVMを行います.
PythonとRノードはKNIME extensionでインストールすることができます.
今回は,「R Predictor/Learner」と「Python Predictor/Learner」を使用しました.
Rについては何度もこれまでの記事で紹介していますが,PythonをKNIMEで処理する際にはいくつか事前の設定が必要です.
How to Setup the Python Extension | KNIME
KNIME Python Integration Installation Guide
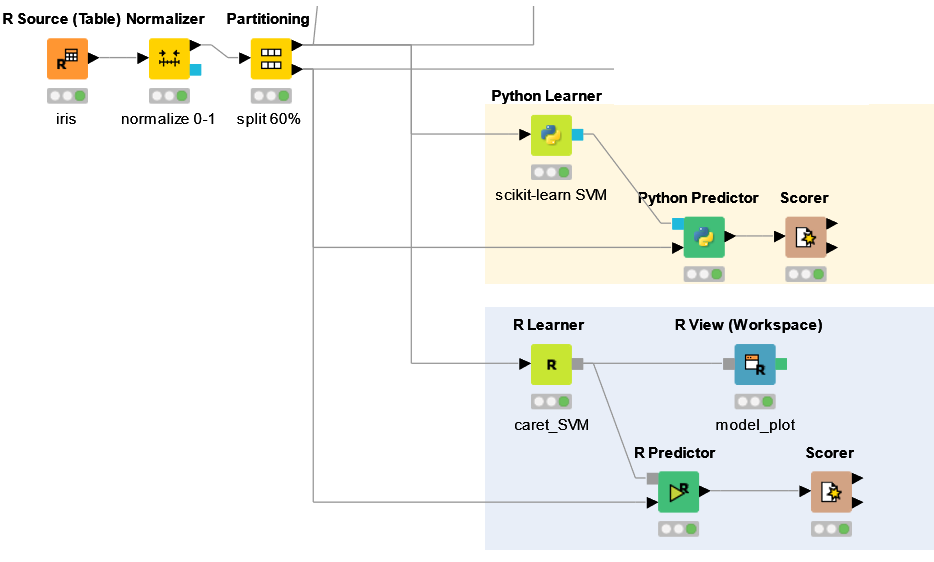
「R Learner/Predictor」を使ったWorkflow
Rのcaretパッケージを使ったSVMについては下記記事を参考にしました.
Rによる機械学習:caretパッケージの使い方 | Logics of Blue
The caret Package
【R】SVMの基礎
まず,「R Learner」でグリッドサーチと交差検証を行い,最適なパラメーターを見つけます.その後得られたパラメーターを用いて学習データからモデルを作成します.
#R Learner library(caret) library(rlang) library(e1071) library(doParallel) cl <- makePSOCKcluster(4) registerDoParallel(cl) data <- knime.in set.seed(0) irisSVM <- train( Species ~., data = data, method = "svmRadialSigma", tuneLength =10, tuneGrid = expand.grid(C=seq(1,10,1), sigma=seq(0.1, 2, 0.1)), trControl = trainControl(method = "cv") ) knime.model <-irisSVM
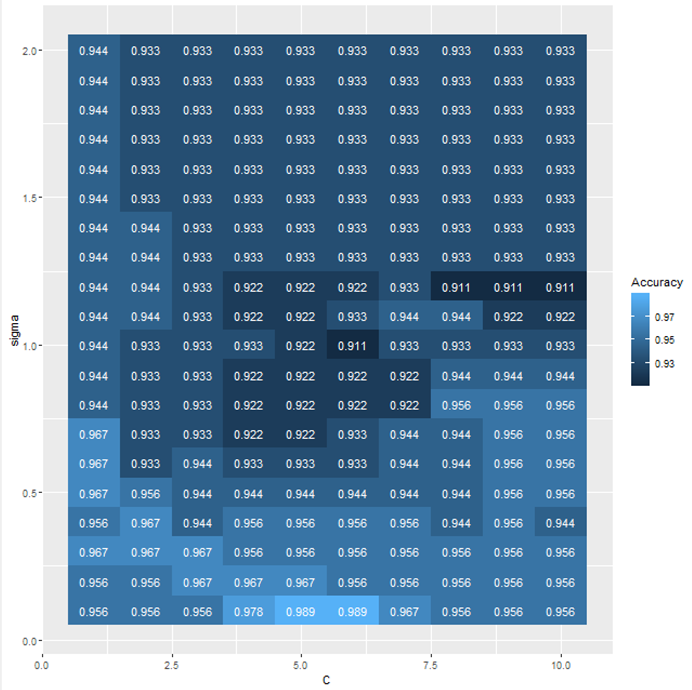
パラメーターの最適化について可視化するために「R Views」を用いました.
parameter_CとsigmaによってAcuracyがどう変化するかを可視化します.
# R Views library(ggplot2) library(gridExtra) library(data.table) model <- knime.model model_plot <- ggplot(model$results, aes(x = C, y = sigma, fill=Accuracy)) + geom_tile() + geom_text(aes(label = round(Accuracy, digits = 3)), color = "white") #geom_point(aes(colour = Accuracy), size = 8) + #scale_colour_gradient(low = "#0068b7", high = "#f39800") #scale_x_log10() model_plot
実行結果を以下に示します.
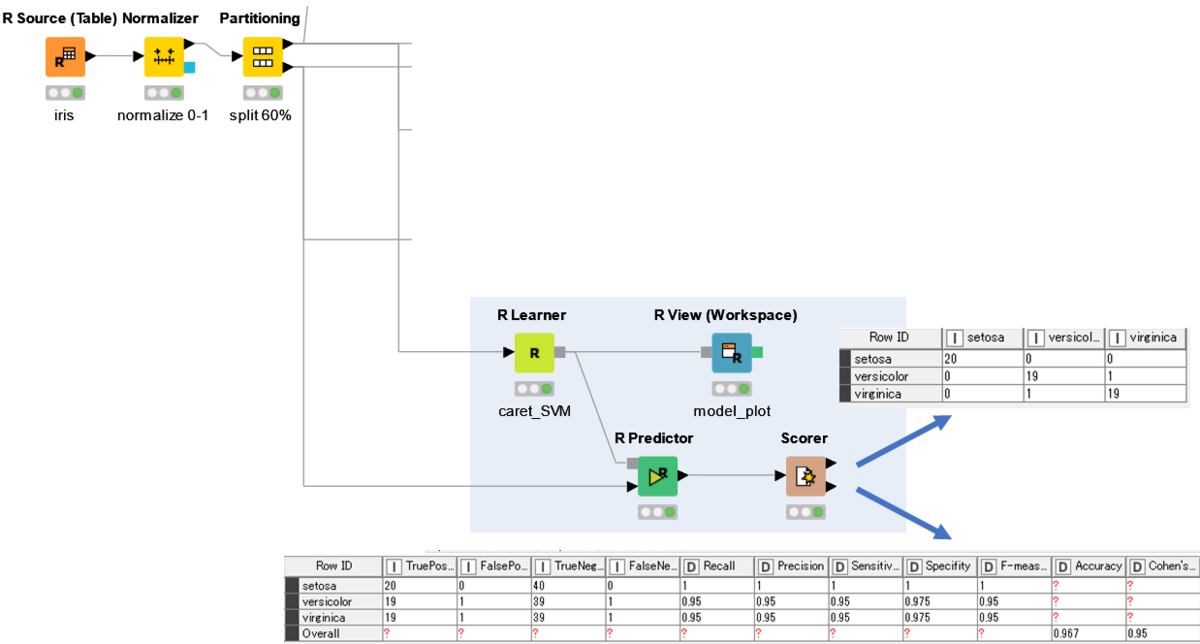
続いて,「R Predictor」でテストデータの予測を行います.
predIrisSVM <- predict(knime.model,knime.in) predIrisSVM knime.out <- cbind(knime.in, predIrisSVM)
最後に「Scorer」で結果を出力します.
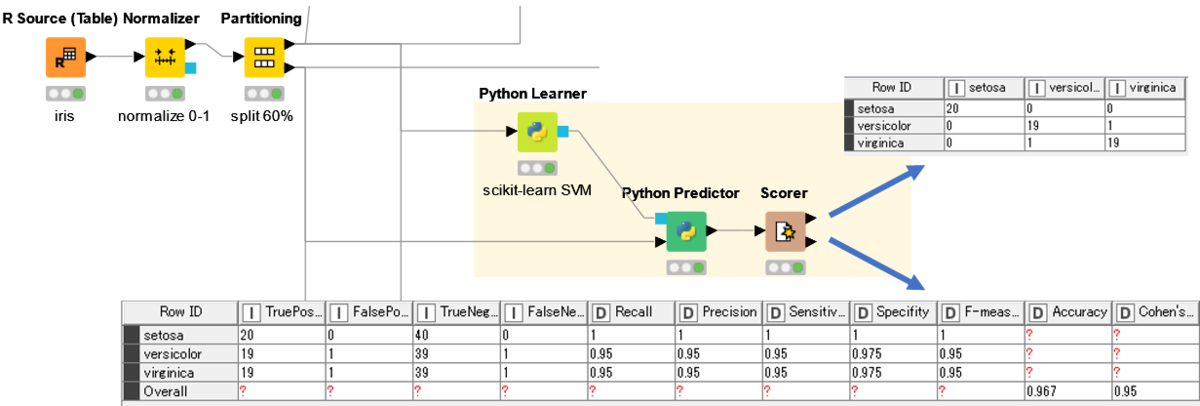
「Python Learner/Predictor」を使ったWorkflow
続いて,Pythonのscikit-learnを使ってSVMを行います.
前編でも紹介した参考書と,以下の記事を参考にしました.
[機械学習] iris データセットを用いて scikit-learn の様々な分類アルゴリズムを試してみた - Qiita
scikit-learnのSVMでirisデータセットを分類 | note.nkmk.me
「Python Learner」の結果を以下に示します.
# Python Learner import matplotlib.pyplot as plt import pandas as pd import mglearn from IPython.display import display from sklearn.model_selection import train_test_split from sklearn.model_selection import GridSearchCV from sklearn.svm import SVC iris_data = input_table._get_numeric_data() iris_label = input_table['Species'] param_grid = {'C': [0.001, 0.01, 0.1, 1, 10, 100], 'gamma': [0.001, 0.01, 0.1, 1, 10, 100]} print("Parameter grid:\n{}".format(param_grid)) grid_search = GridSearchCV(SVC(), param_grid, cv=10) output_model = grid_search.fit(iris_data, iris_label) print("Test set score: {:.2f}".format(grid_search.score(iris_data, iris_label)))
得られたモデルを用いて,「Python Predictor」で解析を行います.
#Python Predictor import pandas as pd test_data = input_table._get_numeric_data() test_label = pd.DataFrame(input_table['Species']) test_label.index = range(60) print(test_label) SVM = input_model pred = pd.DataFrame(SVM.predict(test_data)) print(pred) results = test_label.join(pred) output_table = results
最後に「Scorer」で結果を出力します.
KNIMEノードやRノードと同様に精度よく分類することができました.
まとめ
KNIMEで行うSupport Vector Machineについて,2回に分けてご紹介しました.
今回はテストデータが非常に簡単なものでしたが,実際にはもっと複雑なデータに対してSVMを適応していきます.
パラメーターの最適化もRやPython,KNIMEノードそれぞれで少しずつ異なっている部分はあると思うので,それぞれ好みに合わせて選んでよいと思います.
今回は,前回同様グリッドサーチでパラメーターの最適化を行っていきましたが,Pythonなら「optuna」が良いのでは,と勧められたので,今後勉強していきます.