こんばんは,@PKです.
以前の記事では,Random Forestについて3回に分けてご紹介し,機械学習の基礎的な手法(テストデータ・学習データの分類,交差検証,パラメーター最適化)をKNIME Workflowで紹介しました.
【KNIME】〈前編〉KNIMEでRandom Forest:「Random Forest」ノードを使ったWorkflowの紹介 - t_kahi’s blog
【KNIME】〈中編〉KNIMEでRandom Forest:「Parameter Optimization」と「Cross Validation」の実行 - t_kahi’s blog
上記で学んだ手法を踏まえて,今日は機械学習の一つである,Support Vector Machine (SVM) による判別分析のWorkflowをご紹介します.
これまではKNIME専用ノードとR言語を使った処理を行うRノードの両方を紹介してきましたが,今回からPython言語を使ってPythonノードでも,同様の処理を紹介したいと思います.
最近実務でも機械学習を使い始めたことから,「Pythonではじめる機械学習」という本を先輩から勧められて読み始めました.
Pythonのscikit-learnでの機械学習手法についてまとまっていますが,考え方や実務に応用に直結する内容書かれているので,まだ読まれていない方は是非一読するとよいと思います.
KNIMEだけで機械学習をしている方も,考え方を知る上では非常に良い本だと思います.
Pythonではじめる機械学習 ―scikit-learnで学ぶ特徴量エンジニアリングと機械学習の基礎
それでは本題のKNIMEでSVMの紹介をしたいと思います.
Support Vector Machine (SVM)について
サポートベクターマシン(英: support vector machine, SVM)は、教師あり学習を用いるパターン認識モデルの一つである。分類や回帰へ適用できる。1963年に Vladimir N. Vapnik, Alexey Ya. Chervonenkis が線形サポートベクターマシンを発表し[1]、1992年に Bernhard E. Boser, Isabelle M. Guyon, Vladimir N. Vapnik が非線形へと拡張した。
サポートベクターマシンは、現在知られている手法の中でも認識性能が優れた学習モデルの一つである。サポートベクターマシンが優れた認識性能を発揮することができる理由は、未学習データに対して高い識別性能を得るための工夫があるためである。
サポートベクターマシン - Wikipedia
SVMは上記のように回帰や判別分析に使用できる教師あり機械学習の一種です.
SVMについては先ほど紹介した「Pythonではじめる機械学習」やその他様々な記事で紹介されています.
機械学習入門~ハードマージンSVM編~ - Qiita
非線形SVMとscikit-learnによる実装 - Qiita
サポートベクターマシン(support vector machine:SVM)の基礎 - HELLO CYBERNETICS
サポートベクターマシンとは[カーネル法による非線形サポートベクターマシン] - verum ipsum factum
サポートベクトルマシンの考え方 | Logics of Blue
SVMが何なのか,という定性的な理解は上記資料を見ればわかると思います.
今回はirisデータを用いてSpeciesの予測を,SVMを使って行うWorkflowを紹介します.
KNIME Workflow概要
今回ご紹介するWorkflowを以下に示します.
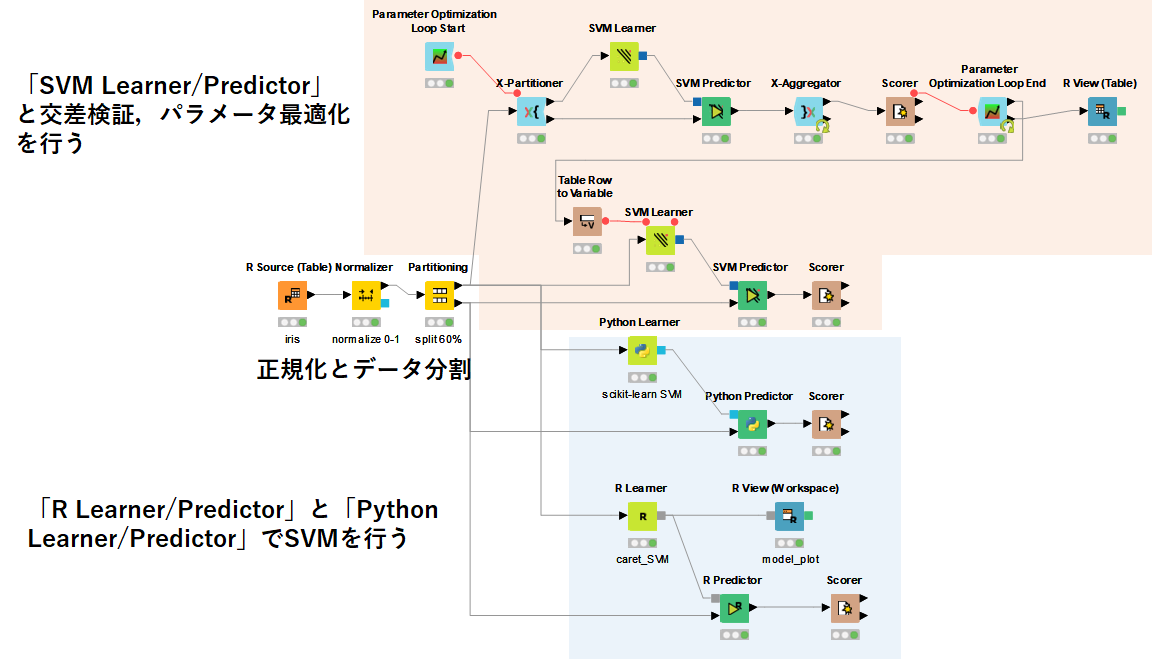
まず,いつも通り,irisデータを読み込んだ後に,「Normalizer」による正規化と「Partitioning」で学習・テストデータ分割を行います.
その後,大きく2つに分かれてSVM解析を行います.
1つはKNIMEの専用ノードである「SVM Learner/Predictor」を使って,交差検証やパラメータ最適化,SVMのモデル構築を行って,実際のテストデータを解析する手法を紹介します.
2つ目は,RとPythonをそれぞれ使ってKNIME上でSVMを実行するWorkflowを紹介します.
Rはcaretパッケージを,Pythonはscikit-learnライブラリを用いてSVMを行います.
irisデータの前処理について
簡単に,irisデータ処理の流れをご紹介します.
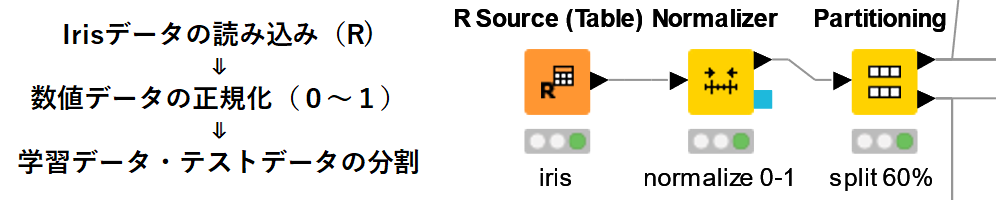
irisデータは「R Source」で読み込みました.
knime.out <- iris
続いてirisデータ内の"sepal length", "sepal width", "petal length",
"petal width"の数値データを「Normalizer」で正規化しました.
前回のRandom Forestは正規化処理はいらないモデルでしたが,SVMに関しては正規化したほうが精度が上がると言われていますので,"Min-Max Normalization"で正規化しました.
最後にデータを学習データとテストデータに分割します.
こちらは「Partitioning」で種のデータがそれぞれ均等になるように分けました.
詳細は以前の記事で紹介しているので参考にしてください.
【KNIME】決定木 (decision tree) をKNIME Workflowで行う - t_kahi’s blog
「SVM Learner/Predictor」を使ったWorkflow
Workflowの流れを以下に示します.
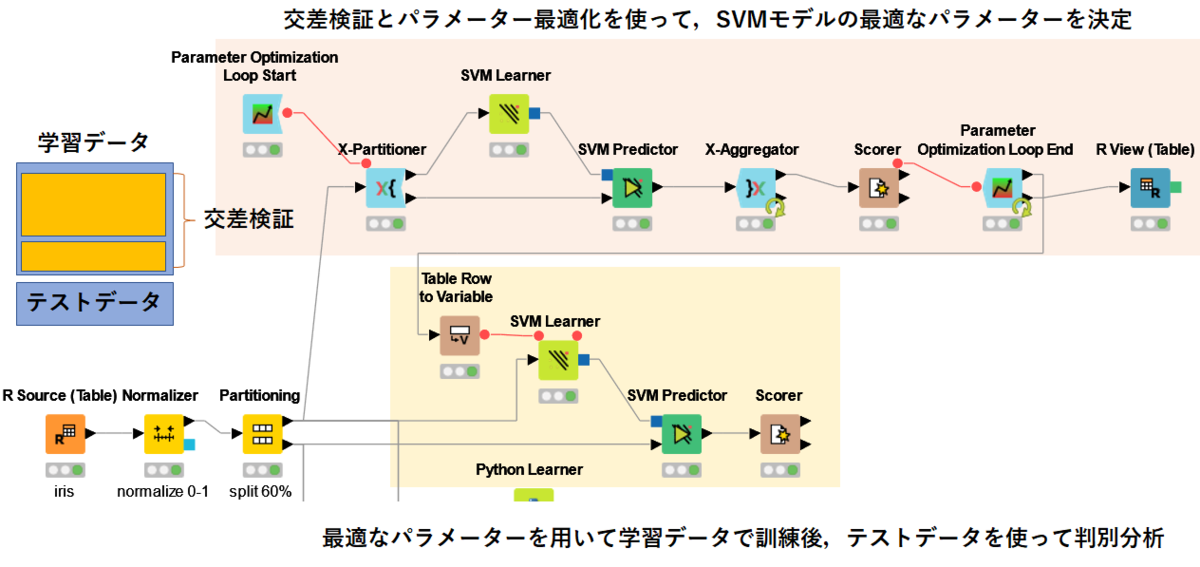
図の上側のデータ解析の流れは以下の通りです.
- 学習データを「X-Partitioner/Aggregator」を用いて交差検証用(10-fold cross validation)の学習データ・テストデータに分割する
- 「SVM Learner/Predictor」をセットしてSVMのモデル(今回はRBF kernel)を設定
- 「Parameter Optimizatio Loop Start/End」でSVMのパラメータ(Cとsigma)のサーチ範囲を設定
- 「Scorer」でAccuracyを表示して,各パラメーターごとのAccuracyを収集
- Accuracyが高いパラメータセットを選択する
また,図の下側は,取得したパラメーターを「Table Row to Variable」で変数にし,それを用いて学習データでモデルを作り,実際のテストデータで判別分析を行います.
パラメータ最適化と交差検証については,Random Forestで紹介しましたので,そちらを参考にしてください.
【KNIME】〈中編〉KNIMEでRandom Forest:「Parameter Optimization」と「Cross Validation」の実行 - t_kahi’s blog
「SVM Learner」
まず,「SVM Learner」ノードについて少し触れたいと思います.
nodepit.com
設定画面を以下に示します.
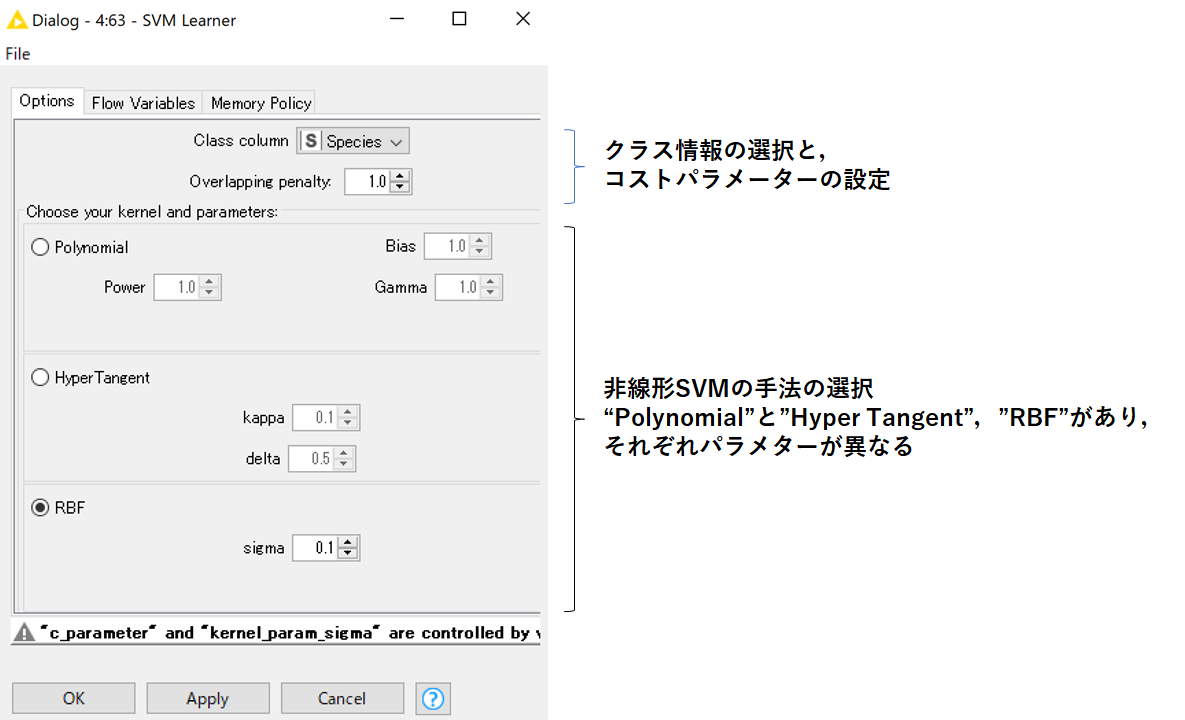
KNIMEのSVMノードは非線形SVMしか選択することができません.
非線形SVMの手法については3つ用意されており,それぞれ設定するパラメーターが異なります.
今回は一般的に使用されていることが多い,RBF kernelを選択しました.
設定するパラメーターは,コストパラメーターである"Overlapping Penalty"と,"sigma"です.
「Parameter Optimizatio Loop Start」
この2つのパラメータを「Parameter Optimizatio Loop Start」でグリッドサーチを行います.
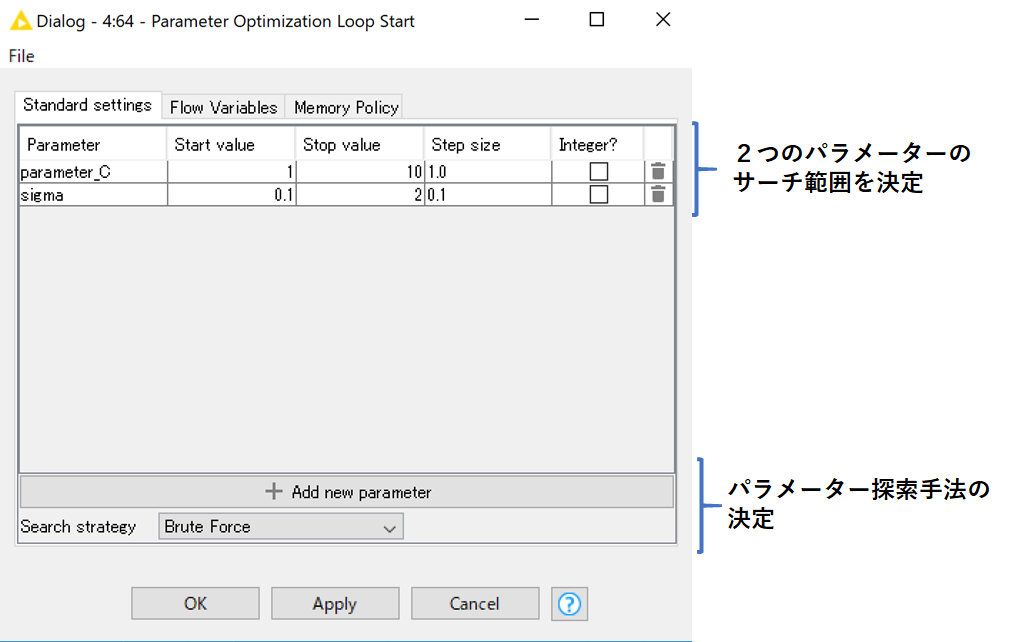
一般的にはSVMのコストパラメーターとかは指数で範囲設定するのですが,このノードではそれができない(悲しい)ので,とりあえず範囲を設定しています.
パラメーター最適化を実行した結果を下図で示します.

SVMの実行は「SVM Predictor」で行います.
作成したモデルとがテストデータを繋げて実行するだけで,結果が出力されます.
nodepit.com
各パラメーターを変えたときの交差検証の結果が「X Aggregator」で出力され,その時のAccuracyが「Scorer」から得られます.
あとはパラメーターを変えながら,Accuracyを出力し,一番スコアが良かったパラメーターとその時のAccuracyを出力します.
「R Views」によるパラメーター最適化の可視化
パラメーターを変えた際にどのようにAccuracyが変化したかを確認したいので,「Parameter Optimizatio Loop End」から出力される"All Parameters"の結果を「R View」を使用して可視化します.
今回はすべての組み合わせを試していることが,図からもわかると思います.
# R VIews library(ggplot2) library(gridExtra) data <- knime.in data$Step <- seq.int(nrow(data))#rowの追加 colnames(data)[3] <- "Accuracy" data_accuracy <- ggplot(data, aes(x = Step, y =Accuracy)) + geom_line(colour="blue") data_parameter_C <- ggplot(data, aes(x = Step, y =parameter_C)) + geom_line(colour="#D55E00") data_sigma <- ggplot(data, aes(x = Step, y = sigma)) + geom_line(colour="#E69F00") library(gridExtra) grid.arrange(data_accuracy, data_parameter_C, data_sigma, ncol = 1, nrow = 3)

「Table Row To Variable」でパラメーターを変数にしてSVM
最適化したパラメーターを使って,テストデータの解析を行います.
以下に示すように,「Table Row To Variable」でパラメーターを変数に変換します.
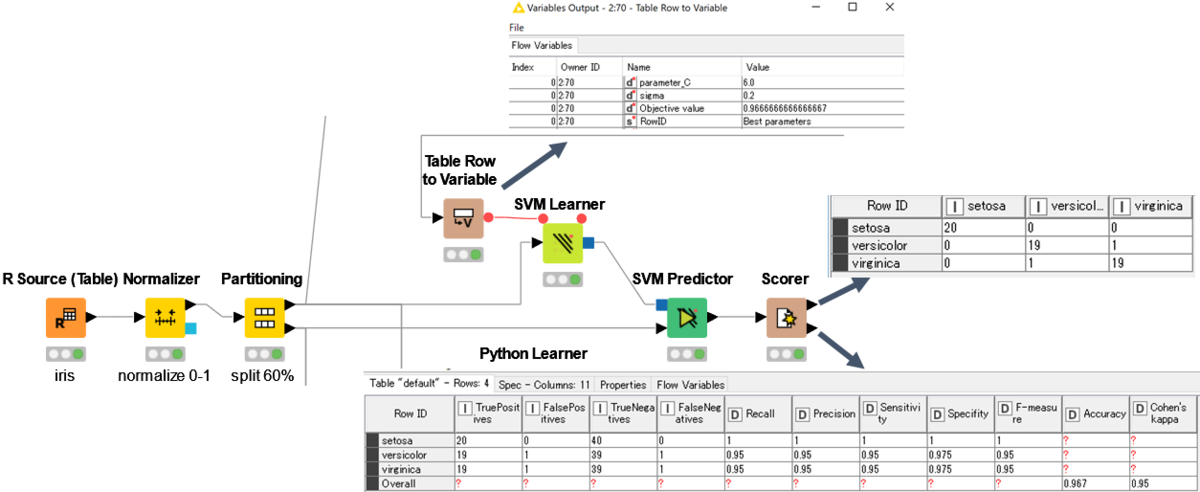
この変数を使って,「SVM Learner」のパラメーターを設定し学習データを使ってモデルを作成します.
その後,「SVM Predictor」でirisの種を予測し,結果を「Scorer」で出力しました.
上図のとおり,”Accuracy”=0.967となりました.
まとめ
KNIME WorkflowでSVMを行う手法を紹介しました.
パラメーターの最適化手法については,今回は総当たりでパラメーターを探しましたが,もう少しいい方法があると思うので,引き続き調べていきます.
次の〈後編〉でRとPythonを使ったWorkflowの説明をします.
最後に今回のWorkflowの動画を紹介します.
youtu.be