こんにちは,@PKです.
今日は判別分析の一種である線形判別分析(Linear Discriminant Analysis)のKNIME Workflowについて紹介したいと思います.
- 線形判別分析(Linear Discriminant Analysis) とは
- KNIME Workflowの概要
- 「Linear Discrimination Analysis」による線形判別分析
- Rノードによる線形判別分析
- まとめ
- おまけ
線形判別分析(Linear Discriminant Analysis) とは
判別分析??という方もいらっしゃると思うので,少しだけ紹介します.
判別分析(はんべつぶんせき、英: discriminant analysis)は、事前に与えられているデータが異なるグループに分かれる場合、新しいデータが得られた際に、どちらのグループに入るのかを判別するための基準(判別関数)を得るための正規分布を前提とした分類の手法。
判別分析 - Wikipedia
判別分析は,グループの情報が与えられたときに,それらをうまく分離する分析手法のことで,今回の線形判別分析は,超平面・直線による判別を指すようです. (*線形判別分析は等分散性が必要)
線形判別分析(Linear Discriminant Analysis, LDA)~多クラスにも応用できる線形クラス分類~ データ解析・マイニングとR言語
今回はこの線形判別分析をKNIME Workflowで行う手法について,具体例を出しながら紹介していきたいと思います.
KNIME Workflowの概要
今回ご紹介するWorkflowは以下の通りです.
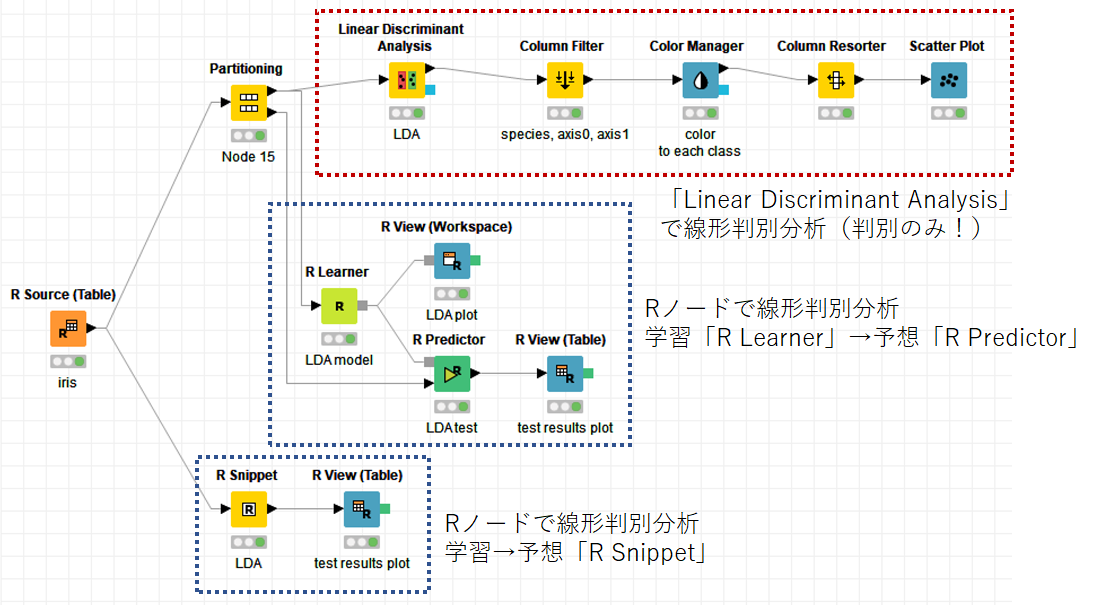
サンプルデータとしてirisを使って,種ごとの判別分析を行います.
#R Table knime.out <- iris head(iris)
> knime.out <- iris + head(iris) Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 2 4.9 3.0 1.4 0.2 setosa 3 4.7 3.2 1.3 0.2 setosa 4 4.6 3.1 1.5 0.2 setosa 5 5.0 3.6 1.4 0.2 setosa 6 5.4 3.9 1.7 0.4 setosa
KINMEの線形判別分析を行うノード「Linear Discrimination Analysis」を使った判別分析についてと,RノードでRのlda関数を用いた線形判別分析の2種類をご紹介します.
「Linear Discrimination Analysis」による線形判別分析
KNIME Workflowには線形判別分析を行う「Linear Discrimination Analysis」というノードがあります. nodepit.com
この「Linear Discrimination Analysis」を使った判別分析のKNIME Workflowも公開されており,誰でも使うことができます.
How to use the Linear Discriminant Analysis – KNIME Hub
しかし,現状では「Linear Discrimination Analysis」に学習→予測の機能が付いていません.
new LDA node - KNIME Extensions - KNIME Community Forum
上記フォーラムでも
The LDA node currently only does dimensionality reduction, no classification. Think of it as PCA that takes into account the class information.
となっているため,あくまでもクラス情報を加味したPCAとして使うことしかできないようです.
せっかくなので判別分析をしてみましょう.
予測ができないので分ける必要はないのですが,「Partitioning」でそれぞれの種ごとに分けて(左下図),その後「Linear Discriminant Analysis」の設定へと進みます(右下図)
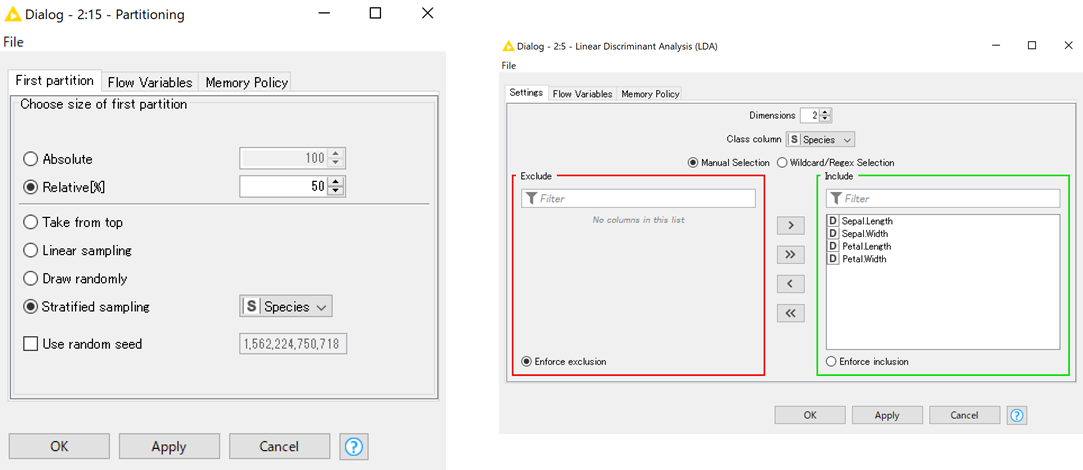
「Linear Discriminant Analysis」の設定では次元数や判別分析を実行するカラム,グループのカラムを指定して実行します.
以下に実行結果の表の一部を示しています.
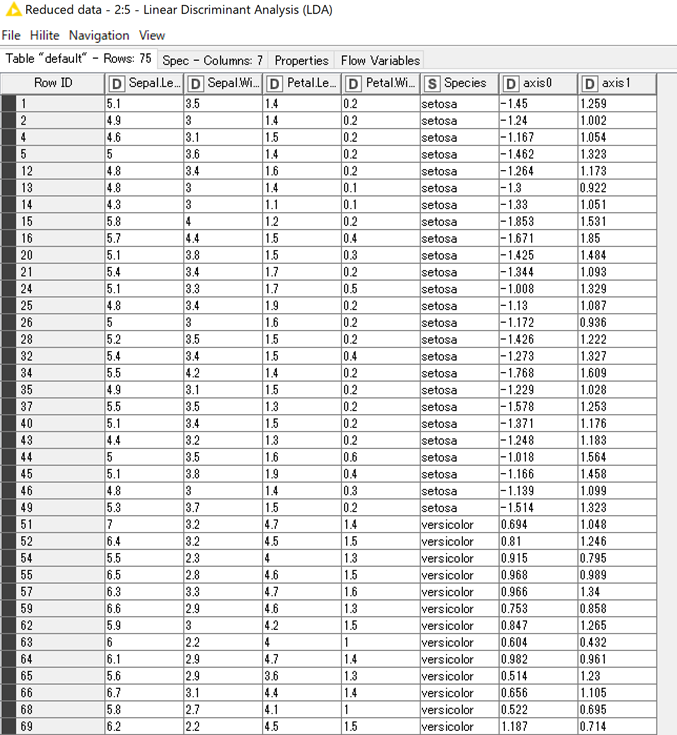
右側のAxis0, Axis1が判別分析の判別係数を表しています.
この後は,得られたデータを可視化するための簡単な処理が続きます.
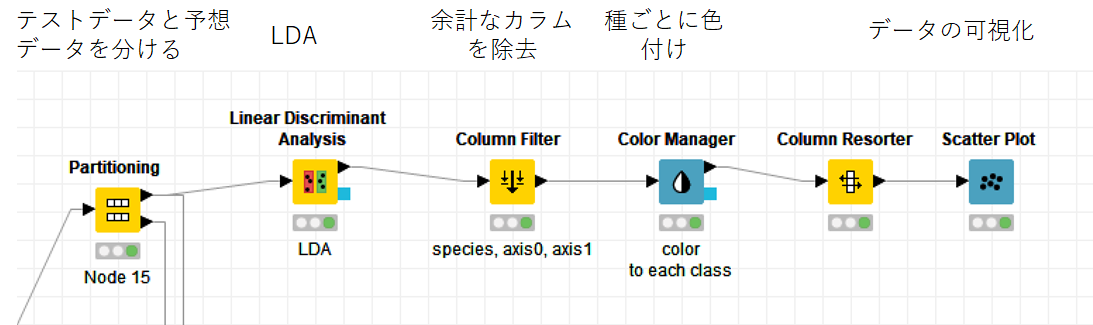
まずは可視化に不必要なからむを「Column Filter」で除いて,「Color Manager」で種ごとに色を付けます.
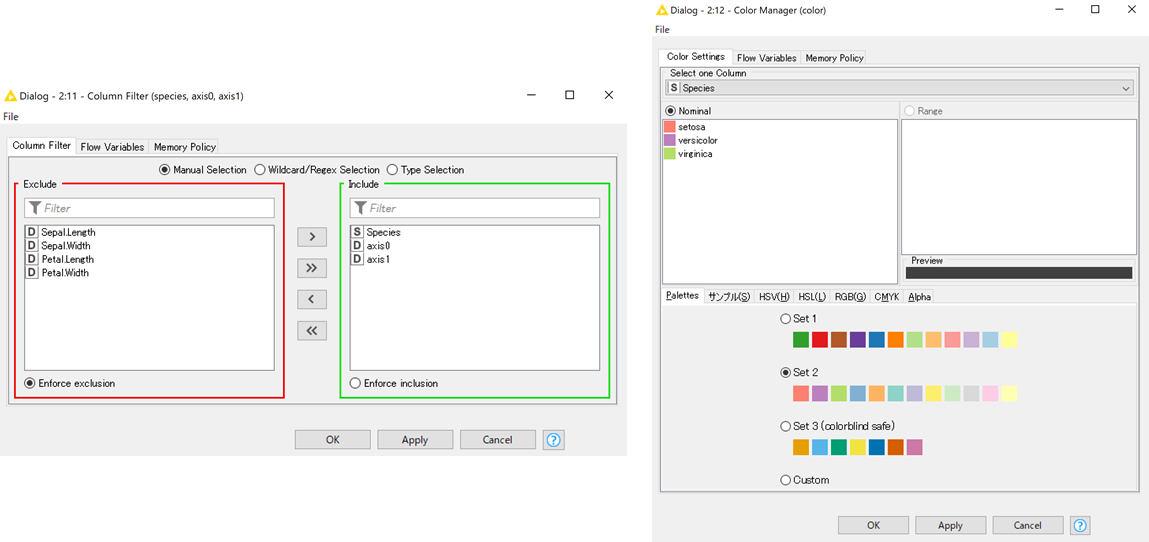
「COlumn Resorter」でカラムの順序を変えて(Scatter Plot表示の時に少しだけ設定を楽にするため),実行をすると右下図のような色付けされた表が出力されます.
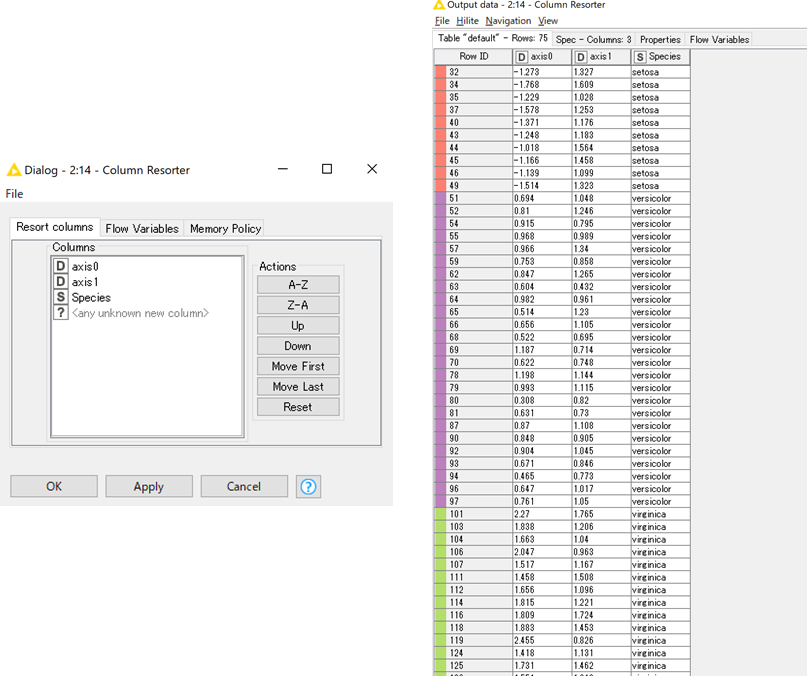 あとはこのデータを「Scatter Plot」で表示すれば完成です.
あとはこのデータを「Scatter Plot」で表示すれば完成です.
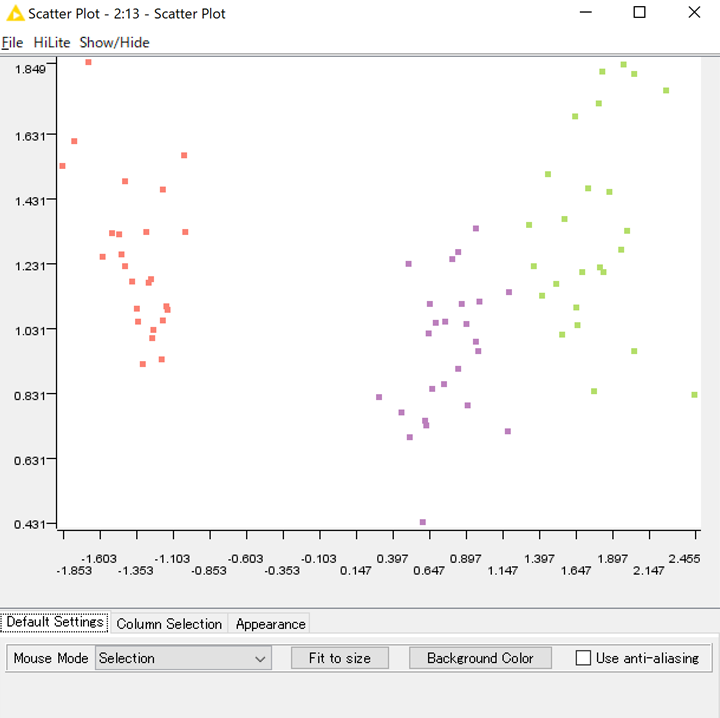
"versicolor","virginica"は距離が近いのでご判別される可能性があります.
とはいえKNIMEの「Linear Discrimination Analysis」では肝心の予測ができないので,Rノードを使ったWorkflowの説明をしたいと思います.
Rノードによる線形判別分析
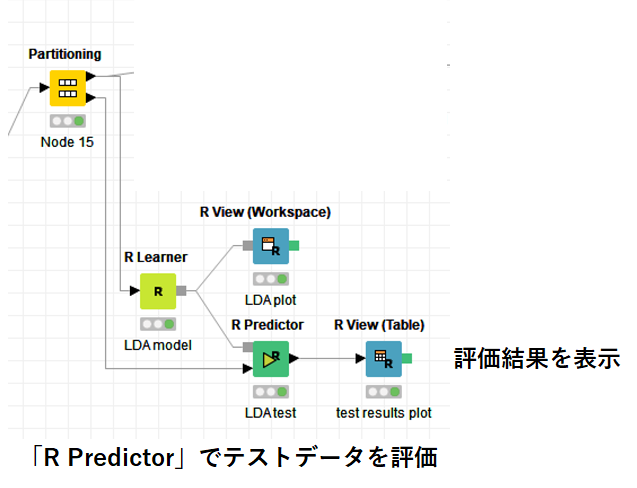
KNIMEのノードでは学習→予想ができないことが分かったので,ここからが本番です.
以下にRノードWorkflowをお示しします.
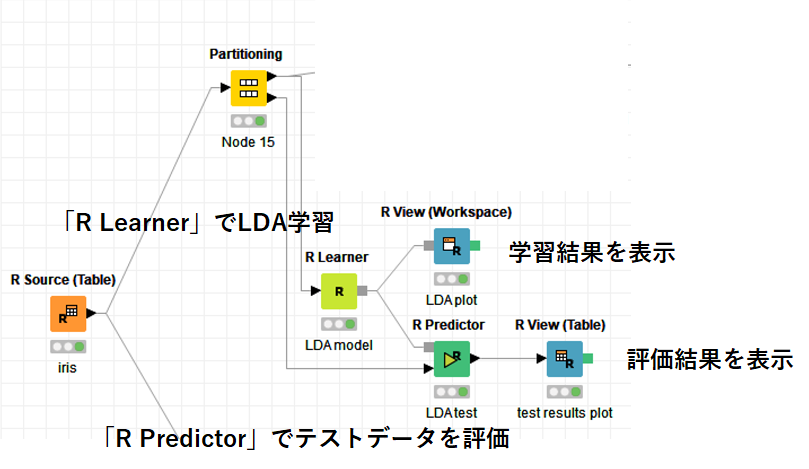
今回使った主なRノードは「R Learner」と「R Predictor」です.
nodepit.com
nodepit.com
「R Learner」ではRスクリプト+モデルを作成することができます.
それを「R Predictor」に受け渡して,テストデータで実際の判別分析を行ってみます.
「R Learner」のRスクリプトは以下の通りです. MASSパッケージのlda関数を使って線形判別分析を行います.
#R Learner library(MASS) train <- knime.in iris.lda<- lda(Species~ .,train)# 線形判別分析の実行 iris.lda#結果の確認 table(train[,5],predict(iris.lda)$class)#学習データの判別結果確認 knime.model <- iris.lda
実行した際に判別ができているかどうかを確認しておきます.
以下の通り,判別分析関数と,"setosa","versicolor","virginica"がそれぞれきれいに分類されていることが最後のテーブルで確認できます.
# R Learner console Call: lda(Species ~ ., data = train) Prior probabilities of groups: setosa versicolor virginica 0.3333333 0.3333333 0.3333333 Group means: Sepal.Length Sepal.Width Petal.Length Petal.Width setosa 5.064 3.448 1.492 0.252 versicolor 6.024 2.780 4.252 1.320 virginica 6.596 3.008 5.560 2.080 Coefficients of linear discriminants: LD1 LD2 Sepal.Length 0.8764488 -0.3374557 Sepal.Width 1.4133696 2.1524568 Petal.Length -1.8823954 -1.1211685 Petal.Width -3.3802799 3.4671372 Proportion of trace: LD1 LD2 0.9869 0.0131 setosa versicolor virginica setosa 25 0 0 versicolor 0 25 0 virginica 0 0 25
上記結果から,うまく3つの種を判別できているようです.
判別分析の判別係数の結果を先ほどの「Linear Discrimination Analysis」と同じように表示させたいときはモデルのデータを可視化するために「R View (workspace)」を使います.
LDA関数の結果をプロットするのは以下を参考にしました.
r - Linear discriminant analysis plot using ggplot2 - Stack Overflow
「R View」のスクリプトは以下の通りです.
#R VIew (workspace) library(ggplot2) iris.lda <- knime.model irisProjection <- cbind(scale(as.matrix(iris[,-5]),scale=FALSE) %*% iris.lda$scaling,iris[,5,drop=FALSE]) irisProjection$row_num = 1:nrow(irisProjection) p <- ggplot(data=irisProjection, aes(x=LD1,y=LD2,col=Species)) + geom_point() + geom_text(aes(label = row_num)) p
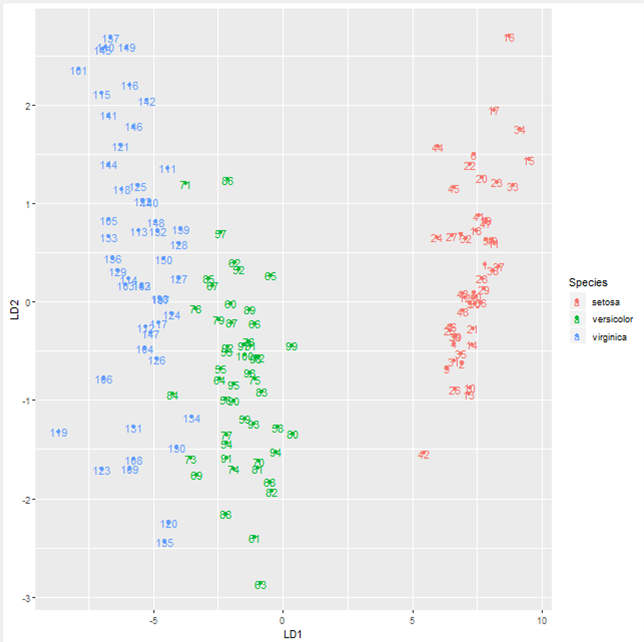
ここまでで,学習データはうまくいっていることが確認できたので,「R Peredictor」で実際のテストデータを判別していきます.
先ほど「Partitioning」で分けた半分のテストデータを判別できるか検証していきます.
「R Predictor」のスクリプトは以下の通りです.
# R Predictor iris.lda <- knime.model test <- knime.in Species <- knime.in$Species iris.test<-predict(iris.lda,test) table(test[,5],iris.test$class)#判別結果の確認 knime.out <- data.frame(iris.test$x,iris.test$class,Species)
モデルの読み込みは"knime.model"だけで良いので意外と簡単で便利です!
判別結果を確認します.
setosa versicolor virginica setosa 25 0 0 versicolor 0 23 2 virginica 0 3 22
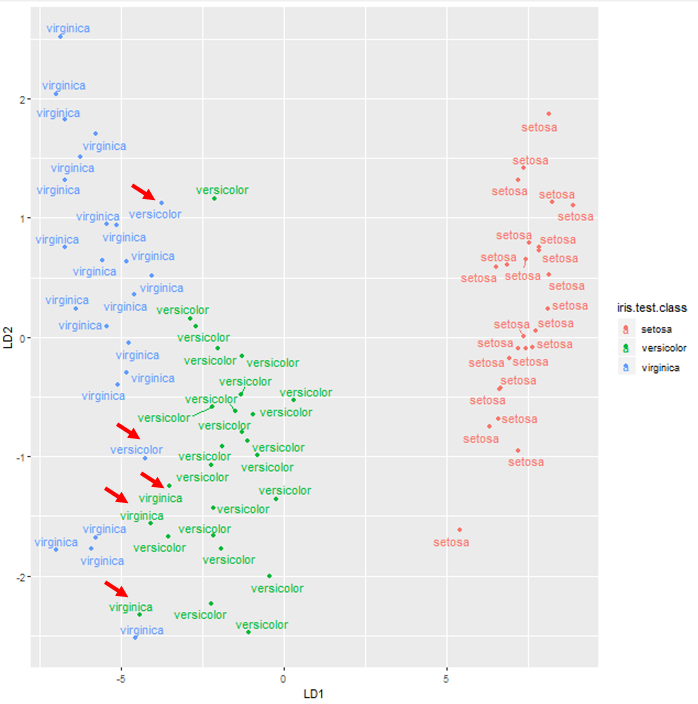
versicolorとvirginicaは先ほどの学習結果からも距離が近いので誤判別が起きています.
テストデータの判別関数値の散布図を示します.
この時,色で判別テスト結果を表示し,ラベルで元の種情報を表示します.
ラベル情報の付加は以下の記事を参考にしました.
ggplot2 パッケージによる可視化の際のラベルの重なりを防ぐ|Colorless Green Ideas
「R View」の結果は以下の通りです.
library(ggplot2) library(ggrepel) data <- knime.in ggplot(data, aes(x = LD1, y = LD2, color = iris.test.class, label = knime.in$Species)) + geom_point()+ geom_text_repel()
赤字で示した部分が誤判別をしている部分です.
まとめ
線形判別分析をKNIME Workflowで行いました.
残念ながらKNIMEのノードでは学習→予測を行うことができないので,Rノードを使うのが良いかもしれません.
この線形判別分析は、各グループの母分散が等しいという仮定(等分散性)に基づいているので,分散が異なる場合は違う判別分析を試したほうがいいでしょう.
Rでは様々な判別分析手法があるらしく,他手法も含めて今後比較していきたいです.
Rで判別分析いろいろ(11種類) - Qiita
最後にWorkflow全体の動画を示します.
youtu.be
おまけ
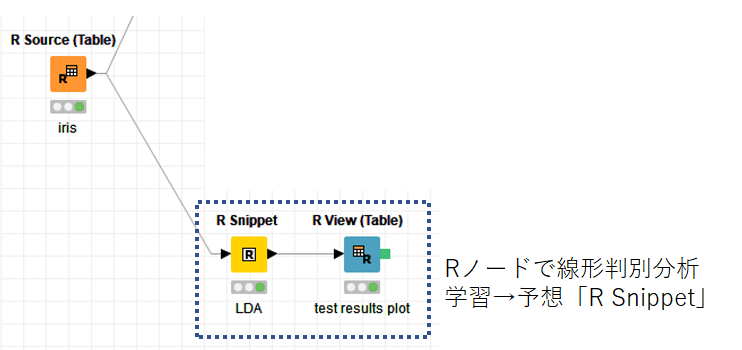
実は「R Learner」とか「R Predictor」など使わなくても「R Snippet」で完結できます.
「R Source」→「R Snippet 」→「R View」なので,もうKNIMEでやる意味が一切ないですね…
# R Snippet library(MASS) data.iris <- knime.in nr <- nrow(data.iris) row.train <- sample(nr, floor(0.5 * nr)) train <- data.iris[row.train,] test <- data.iris[-row.train,] iris.lda<- lda(Species~ .,train)# 線形判別分析の実行 iris.lda#結果の確認 table(train[,5],predict(iris.lda)$class)#学習データの判別結果確認 Species <- test$Species iris.test<-predict(iris.lda,test) table(test[,5],iris.test$class) knime.out <- data.frame(iris.test$x,iris.test$class, Species)
結果は「R View」で確認します(結果は先ほどと全く同じなので割愛します)