こんにちは,@PKです.
はじめに
以前、KNIMEを使って多変量データの次元を削減したいと思い、調べていた時に
「Seven Techniques for Data Dimensionality Reduction 」というタイトルのKNIME official blogとpdf資料を見つけました.
KNIMEのblogとpdf資料では次元削減の7つのテクニックについて紹介されています.
https://files.knime.com/sites/default/files/inline-images/knime_seventechniquesdatadimreduction.pdf
詳細は、上記資料に記載されているのですが、内容をメモしておきます.
サンプルデータは下記「wine.data」を用いました.
Index of /ml/machine-learning-databases/wine
「file reader」でwineのラベルと13の特徴量が読み込みます.

また、特徴量選択のためのノードの多くは、Analytics => Mining => Feature Selectionに含まれています.

Seven Techniques for Data Dimensionality Reduction
Missing Values Ratio
欠損値を多く含んでいる特徴量は意味のない情報であることが多いことから、欠損値を含む割合の閾値を設定し基準以上の欠損値を含んでいる特徴量を削除する手法です.
これは、「missing value column filter」ノードで行うことができます.

上図のように、「missing value column filter」の設定画面で必要なカラムを選択し、missing value threshold (in %)で欠損値の割合の閾値を決めます.この設定値より多く欠損値をもつカラムは除去されます.
(今回のデータセットは欠損値が無かったので意味が無かったですね、、)
Low Variance Filter
データの変動がほとんど無い(分散が小さい)特徴量は重要な意味を持つことが少ない、と考えそのようなカラムを除くことで次元削減を行います.
数値が大きい特徴量の場合、データの変動幅も大きくなります.なので、この手法を使用する際はデータを正規化する必要があります.
まず、「normalizer」で特徴量を正規化します.

その後、「low variance filter」で処理を行います.Variance Upper Boundで、除去する分散の閾値を決めます.
nodepit.com


3つの特徴量が残ったので、それらを「3D scatter plot」ノードで可視化した結果を以下に示します.
各色がカテゴリーを表しています.これだけでも、それぞれのカテゴリーが分離しており、それぞれの特徴量が意味を持っているようです.

High Correlation Filter
特徴量間で似たような変動を示す場合は、それらの特徴量が持つ情報は似ている可能性があります. 相関する特徴量を除くことで、意味のある特徴量だけを残すことができます.
まずは「liner correlation」ノードで線形相関のモデルを作成します.

各特徴量間のcorrelation matrixを以下に示します.
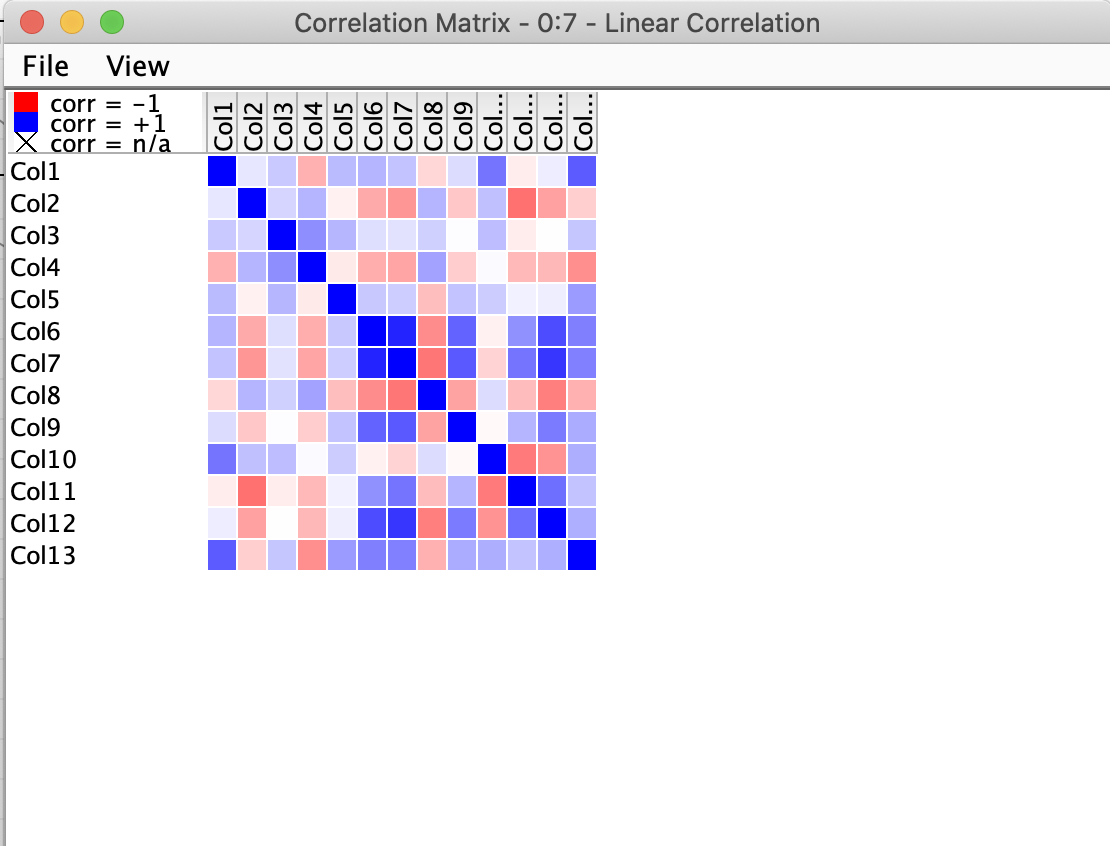
続いて、「correlation filter」で相関する特徴量を除きます.Correlation Thresholdで閾値を決めます.下図の場合は0.5以上の相関係数を持つ重複する特徴量を除き、6つの特徴量に絞り込んでいます

得られた6つの特徴量を3Dプロットした結果を以下に示します.

Random Forests / Ensemble Trees
予めカテゴリーが決められている教師ありの場合は、random forestで評価して属性重要度からどの特徴量が重要かを示すことができます.
今回の例でもwineのカテゴリーがあるので、「random forest Learner」ノードを使って解析を行うことができます.
KNIMEでrandom forestを行う方法については下記ブログで紹介していますので、詳細は割愛します.
属性重要度の高い順に特徴量を表示し、上位3つをプロットした結果を示します.


Principal Component Analysis (PCA)
主成分分析を使ってもデータの次元を落とすことができます.KNIMEでは「PCA compute」というノードを使用します.
この場合もLow Variance Filterと同じく分散が大きい特徴量に引きずられるので、事前にデータを正規化する必要があります.
KNIMEでPCAを行う手法は下記ブログで紹介していますので、こちらも詳細は割愛します.
PCAで次元削減をした結果を以下のようにプロットします.


Backward Feature Elimination & Forward Feature Construction
Backward Feature Eliminationは全ての特徴量を用いて学習を行い、そこから1つずつ特徴量を抜いて、最もモデルの精度がよかった特徴量の組み合わせを選ぶ手法です.
Forward Feature Constructionは、逆に1つの特徴量からスタートし、そこから1つずつ特徴量を組み合わせ最も精度の良い組み合わせを探す手法です.
参考:変数選択(Feature Selection)手法のまとめ - 学習する天然ニューラルネット
冒頭で紹介したように、「Backward Feature Elimination」と「Forward Feature Construction」はFeature selectionのMetanodeに用意されています.

これらは名前の通り、メタノードとして用意されています.

メタノードの中身は、下図のようなノード構成になっています.

デフォルトはNaive Bayesですが、メタノードの中で自分の好きなモデルを組み込むことができます. あとは、Feature selection loopで特徴量を増減させながらモデルの精度を検証していき、最も意味のある特徴量を選択することができます.
メタノードとして用意されているので、自分の好きなモデルで簡単にこれらの手法を試すことができて非常にいいですね・
さいごに
新年度が始まり、年々忙しさが増してきますが、解析手法の勉強も続けていきたいです…!
今回はSeven Techniques for Data Dimensionality ReductionでKNIMEを用いた次元削減の勉強をしました.

適当なサンプルデータを使って自分で触りながら理解していくのも楽しいですが、KNIME Hubという素晴らしいツールがあるので、下記からサンプルWorkflowをダウンロードして触ってみるのもいいですね.