「KNIMEを使ってプレートのアッセイデータを解析する」という内容の後半部分について説明をしたいと思います. 解析といっても,今回は得られたデータをヒートマップで表示するということと,化合物の阻害活性IC50値を算出するということをKNIME上で行います.
KNIMEのフローですと右側の赤点線囲いの部分です.

前半の内容はこちらに記載しました.
前半の最後にお示ししましたが,joinerでアッセイプレートのフォーマットや濃度の情報と,測定結果のデータを各wellごとにマージしたテーブルを以下に示します.

KNIMEのHCS toolを見ていると,Data Viewsの部分に,heat map viewというノードがあったので試しに使ってみました.

このplate heatmap viewrでは,以下のように,どの値をどのように表示するかを選択していきます.

Readouts:どの値を用いてヒートマップを表示するか(今回は,解析結果の数値)
Factors:各wellの情報(format1情報)
Plate:column,rowの情報,プレートのラベル等
結果はpng形式で出力されますので,それをImage Port Writerで出力した結果がこちらです.
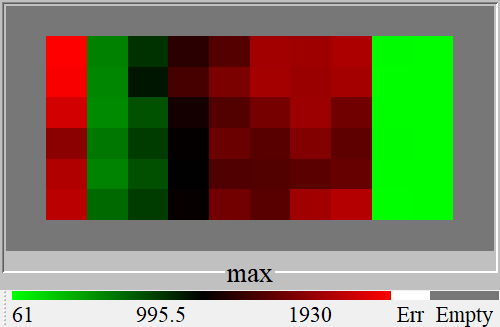 一番左の列がMax値,2列目から各化合物の濃度(n=6)を示しており,左から右に従って濃度が薄くなるにしたがって,阻害値も減少していることがわかります(阻害なので数値は大きくなります).右端の2列は,reference化合物とブランクです.
一番左の列がMax値,2列目から各化合物の濃度(n=6)を示しており,左から右に従って濃度が薄くなるにしたがって,阻害値も減少していることがわかります(阻害なので数値は大きくなります).右端の2列は,reference化合物とブランクです.
このheatmapの活用については今後の宿題にしています...
良いアイデアがあったら是非教えてください.
続いて,得られた化合物の測定データからシグモイドのフィッティングを行いたいと思います.
化合物が入っているwellだけを抜き取りたいので,Row filter ノードを使って化合物が入っている行だけを抽出します.
その後,Dose Response (R)というノードを使用します.
Dose ResponseのNode descriptionを読んでみます.
Calculates and visualizes dose response curves. This is achieved by fitting several non-linear regression models to your data. Measurements are grouped per Compound and Concentration (see Options). Measurements from a zero concentration are used to calculate the fit but are not displayed in the plot because of its logarithmic scale. For a conceptual and technical overview have a look at "Bioassay Analysis using R", Ritz and Streibig, 2005, 5:12, Journal of Statistical Software.
IMPORTANT: This node is using R as back end, make sure that Rserve is running somewhere, and KNIME is accordingly configured (KNIME->Preferences). HCS-Tools Wiki
Nodeの名前の通り,裏でRを使ってシグモイドのフィッティングを行っています. RをインストールせずにこのNodeだけを使うと,こんなエラーが出てきます(何も考えずにポチポチ押してました…).
ERROR Dose Response (R) 3:14 Execute failed: Could not connect to R. Probably, the R-Server is not running. Here's what you need to do: 1) Check what R-server-host is configured in your Knime preferences. 2) If your host is set to be 'localhost' start R and run the following command library(Rserve); Rserve(args = "--vanilla")
ということで,
- Rのインストール
- HCA Tools Wikiを参照しながら,必要なパッケージのインストール
を行います.
Rのインストールに関しては,ちょうどWindowsでのインストール方法に関する記事がありましたので,こちらを参考にしました.
https://qiita.com/FukuharaYohei/items/8e0ddd0af11132031355
また,Rserveというパッケージのインストールについては以下のHCA Tools Wikiを参考にしました.
https://github.com/knime-mpicbg/knime-scripting/wiki/R-server-for-knime#How_to_setup_a_local_RServer
インストールが完了したところで,Dose Responseのdialogを確認します.

上記の図の通り,Parameter of interestの項目に,どの値をつかってフィッティングするのかを選びます.
また,compoundの名前を選択する項目,concentrationの情報を記載する項目にそれぞれ対応する列を選択します.
フィッティングモデルについてですが,descriptionに記載がありました.
Regression Model
The following models (referred to their abbreviation in the configuration dialog) are supported:
LL.3 : Three-parameter logistic
LL.4 : Four-parameter logistic (default)
LL.5 : Five-parameter logistic
W1.2 : Two-parameter Weibull
W1.3 : Three-parameter Weibull
W1.4 : Four-parameter Weibull
4パラメーターロジスティック回帰がデフォルトですし,それで問題ないと思ったので,LL.4を選択しました.
これで解析をした結果は以下の通りです. シグモイド曲線と,統計データがそれぞれ得られるので,pngファイルおよびエクセルファイルで出力をしました.
dose response plot
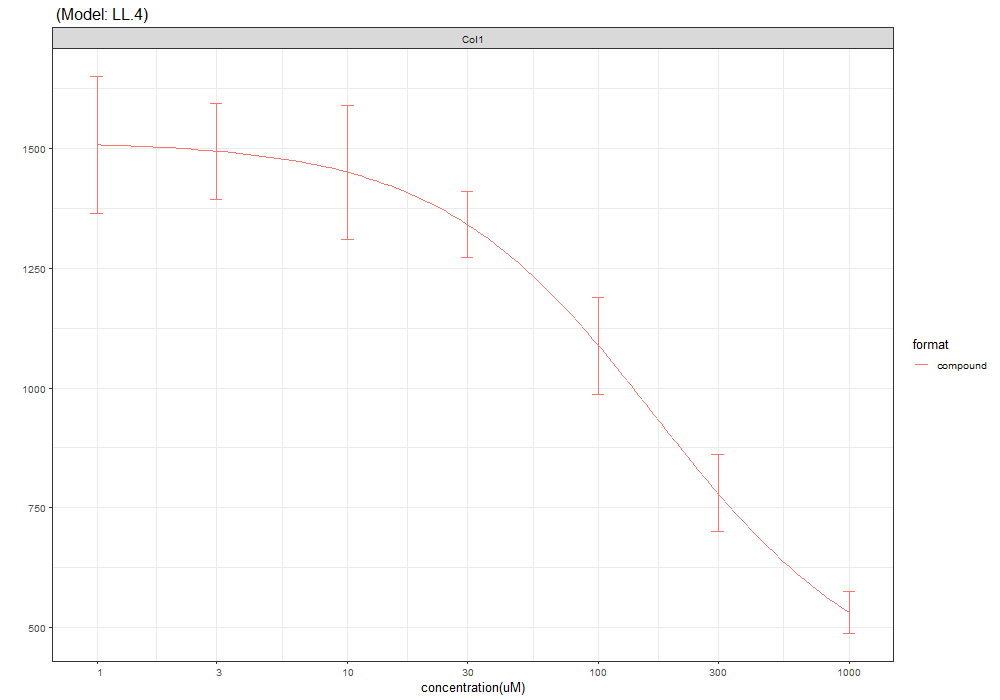
| row ID | format | Response Variable | IC 50 | Std. Error (IC 50) | Lower Limit | Std. Error (Lower Limit) | Upper Limit | Std. Error (Upper Limit) | Slope | Std. Error (Slope) | Symmetry | Std. Error (Symmetry) | Residual standard error | DoF |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | compound | Col1 | 169.4438189 | 51.32286069 | 365.3273459 | 140.6364829 | 1514.176239 | 34.89681933 | 0.999044628 | 0.228391736 | 99.50475587 | 38 |
無事に解析データを取得することができました!
前回自動化した時はpythonを使っていましたが,KNIMEのほうがプログラミング初学者でも簡単にここまでたどり着けるかな…?という気がします(もちろんCUIでできたほうがいいと思ってはいます…).
普段はPrismを使うことが多いのですが,見栄えを気にしなければこちらで充分ですね.
今回は,1化合物のdose response plotとIC50値を取得しましたが,複数ある場合でも同様にまとめてデータを取得することができそうなので,実務でもしっかり使えそうです.
いずれ複数の化合物のデータや,HCAのデータなどを用いてどのように使っていくか,基本の部分をこのブログに書いていきたいと思っています.