こんばんは,@PKです.
今日でこのブログは一周年だそうです. 引き続きKNIMEやCellProfilerについて紹介をしていきたいと思います.
さて,これまでCellProfilerで紹介してきた画像解析は,以下のように,各染色画像が1枚ずつのデータを取り扱ってきました.
しかし,実際のhigh content analysisではアッセイデータを含めて複数の情報と画像データを扱うことのほうが多いです.
この画像とそれに付随する情報(=メタデータ)をまとめて扱うことができるのが,「Metadata」モジュールです.
CellProfilerのMetadataモジュールの概要は以下のサイトから確認できます.
Metadata
今日は,ある画像データセットを題材として「Metadata」モジュールの取り扱いについて,紹介したいと思います.
- 使用する画像データとメタデータについて
- 【KNIME】メタデータの加工
- 【CellProfiler】Imagesモジュール
- 【CellProfiler】Metadataモジュール
- 【CellProfiler】NameAndTypes
- 最後に
使用する画像データとメタデータについて
今回は,以下の論文の画像データを使用させていただきました.
今回の記事ではこの論文の内容には触れませんが,概要としてはU2OS細胞を多重染色し,様々な薬剤を処理した画像から特徴量を抽出してCellProfilerを用いて解析を行っています.
この論文では画像データも公開されている(感動!)ので,この画像データを使って,「Metadata」モジュールを使用しました.
以下のサイトで画像データとメタデータを取得することができます. data.broadinstitute.org
上記サイトは,Broad Bioimage Benchmark Collection (BBBC)と言って,無償ダウンロードができる顕微鏡画像セットが置いてあり研究者は自由に使うことができます.
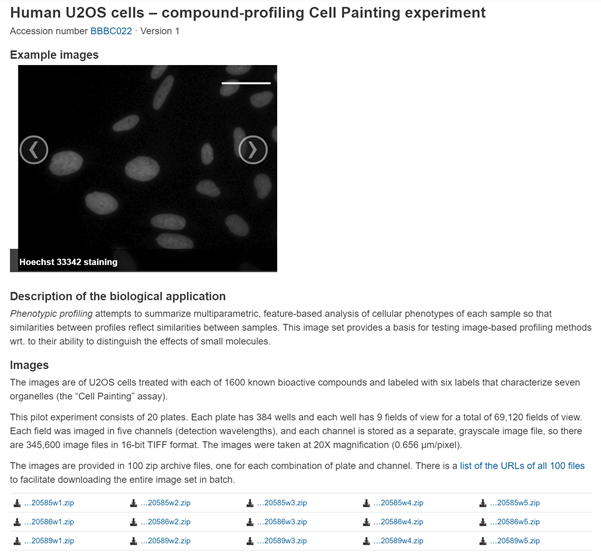
さて,サイトへアクセスすると,以下のように画像セットの説明と大量の画像データのダウンロードリンクがあります.
膨大な画像データなので,細かくファイルが分けられています.
一番上の行の「20585」はプレートNo.を示しています.
また,数字の次の「w1~5」は5つのチャンネルを表しています.
ちなみに,5つの染色については以下論文から抜粋しました.
- Hoechst : Hoechst 33342 (nuclei, blue)
- ER : concanavalin A (ER)
- Syto : SYTO 14 (nucleoli)
- Ph-golgi : phalloidin (actin)&WGA (Golgi)
- Mito : MitoTracker Deep Red (mitochondria)
100のファイルはすべてダウンロードするのは厳しいので,今回は20585w1~20585w5の5つのファイルをダウンロードしました(これだけでも10GB超…).
また,サイトの下にはメタデータのCSVファイルがあります.
このCSVファイルには以下の化合物評価に関する情報が記載されています.

画像データとこのメタデータをダウンロードしたら次のステップに進みます.
【KNIME】メタデータの加工
メタデータの10行を取り出したテーブルを以下に示しています.
一つの行で5つの異なる染色画像データと,プレートの位置情報や化合物の処理条件が紐付けられているようです.
| ImageNumber | Image_FileName_OrigER | Image_FileName_OrigHoechst | Image_FileName_OrigMito | Image_FileName_OrigPh_golgi | Image_FileName_OrigSyto | Image_Metadata_ASSAY_WELL_ROLE | Image_Metadata_BROAD_ID | Image_Metadata_CPD_MMOL_CONC | Image_Metadata_CPD_PLATE_MAP_NAME | Image_Metadata_CPD_SMILES | Image_Metadata_CPD_WELL_POSITION | Image_Metadata_PlateID | Image_Metadata_SOURCE_COMPOUND_NAME | Image_Metadata_SOURCE_NAME | Image_Metadata_Site | Image_Metadata_Time | Image_PathName_OrigER | Image_PathName_OrigHoechst | Image_PathName_OrigMito | Image_PathName_OrigPh_golgi | Image_PathName_OrigSyto |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | IXMtest_A01_s1_w22CA7BDF3-1425-4AE6-8E57-9C004D6F7FF2.tif | IXMtest_A01_s1_w164FBEEF7-F77C-4892-86F5-72D0160D4FB2.tif | IXMtest_A01_s1_w58DA6AF62-1799-4B85-BE17-744E12ADC245.tif | IXMtest_A01_s1_w47457436C-5BE7-4DA2-B966-CCF64B9F0088.tif | IXMtest_A01_s1_w3FD54227A-6BE4-460B-8E2C-54B8709F6E33.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 1 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 2 | IXMtest_A01_s2_w246FFAEE1-BEB6-4C81-913B-B979EC0C4BC3.tif | IXMtest_A01_s2_w1AA6B1894-F561-42EE-9D1D-E21E5C741B75.tif | IXMtest_A01_s2_w50F1562CD-EBCF-408E-9D8D-F6F0FDD746C8.tif | IXMtest_A01_s2_w46657239A-5AFE-4C29-BB9B-49978EFE4791.tif | IXMtest_A01_s2_w3A597237B-C3D7-43AE-8399-83E76DA1532D.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 2 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 3 | IXMtest_A01_s3_w22AD5BD08-58A3-466D-A364-BE116BD98696.tif | IXMtest_A01_s3_w108F3A6FF-A865-40A1-8488-9A5BA31605A9.tif | IXMtest_A01_s3_w5489716D4-2D06-45BE-8D68-F2EEE857929D.tif | IXMtest_A01_s3_w47B5F2FF6-A5A2-4101-AC2A-0A821FDE7445.tif | IXMtest_A01_s3_w30B946780-C923-4A40-8EC5-16262CFF03A6.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 3 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 4 | IXMtest_A01_s4_w231D1EC99-22A8-47A8-9691-691906FA558E.tif | IXMtest_A01_s4_w189967984-D8CB-4449-9CE5-EE2E5F1B8ED4.tif | IXMtest_A01_s4_w5D0566314-B9B2-4299-98AC-9BC33CA94192.tif | IXMtest_A01_s4_w47E34AB5F-23B4-42E1-8B5B-93AB750DA038.tif | IXMtest_A01_s4_w3F582BADD-2249-48D6-BC19-1B4C166A2122.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 4 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 5 | IXMtest_A01_s5_w2B3F07F02-876D-455B-94E4-35A0802022FD.tif | IXMtest_A01_s5_w1DCC72C3D-6ABF-4D49-897E-5F35679A166D.tif | IXMtest_A01_s5_w513201EB0-3500-4A0C-8FAC-9468E5195D1D.tif | IXMtest_A01_s5_w4AFA01300-7373-42C4-872F-0375FA56ECAC.tif | IXMtest_A01_s5_w3EB7ABD45-EE05-41A0-979E-7F3056233176.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 5 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 6 | IXMtest_A01_s6_w27F5A3544-E7D1-4B26-AFD0-416B9F6666B7.tif | IXMtest_A01_s6_w11258626E-836F-4028-A3CB-50B83C60EAFF.tif | IXMtest_A01_s6_w5724C008F-7724-45C3-9175-14CB1B83D10A.tif | IXMtest_A01_s6_w4946663A1-86AC-40DF-943A-4E08B1CD13D9.tif | IXMtest_A01_s6_w3AB6348D8-0D46-488E-A7F5-28C7154735C0.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 6 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 7 | IXMtest_A01_s7_w295CAF1E1-B818-4677-893C-12C7F2DED413.tif | IXMtest_A01_s7_w179C24BDB-EF90-4FE8-BA34-932EA0272FCE.tif | IXMtest_A01_s7_w59DF62B1E-DFA1-43CC-8E5A-E728330183F2.tif | IXMtest_A01_s7_w4F1F709F2-75BD-4C97-8C05-F9DDB5FE4BC9.tif | IXMtest_A01_s7_w31942FB3D-6A47-4BC7-9E29-B1F3A8315EEE.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 7 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 8 | IXMtest_A01_s8_w25D5AAB6A-DAA9-4B11-83B1-3FB83E67C7BC.tif | IXMtest_A01_s8_w1032E967C-5DCF-45C9-A460-2F9FF2234C24.tif | IXMtest_A01_s8_w5DDA6A97F-74B1-4885-B7E2-A45D548546EB.tif | IXMtest_A01_s8_w4A92050CA-2700-43B3-B6C9-DFEDDB126BFB.tif | IXMtest_A01_s8_w32DF8A9F7-6B3A-4CAF-A4F7-6B21886EB183.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 8 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 9 | IXMtest_A01_s9_w296CDDB9B-6E01-4728-808C-B75F055DD8A4.tif | IXMtest_A01_s9_w144EC6EFC-4CAE-411A-B641-EDA50D0E1550.tif | IXMtest_A01_s9_w502337C9E-8FAB-4160-A7DC-03AC77E687D8.tif | IXMtest_A01_s9_w4ED8064A5-A59B-4467-97FF-5D233832619D.tif | IXMtest_A01_s9_w31798561A-387E-4234-B6FF-91E8553A153D.tif | compound | BRD-K98763141-001-06-8 | 8.858401198 | H-BIOA-002-1 | OC(=O)c1cccnc1Nc2cccc(c2)C(F)(F)F | A01 | 20585 | NIFLUMIC ACID | Biomol International Inc. | 9 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
| 10 | IXMtest_A02_s1_w2F2F58829-F3E0-44C8-9F34-A22F20F6005B.tif | IXMtest_A02_s1_w17C9F8BDB-79F0-4F8B-852B-71161631F236.tif | IXMtest_A02_s1_w574184257-3FB3-459B-9FBE-D5781F590253.tif | IXMtest_A02_s1_w48D2F4423-35BE-4871-AF40-3073075D8835.tif | IXMtest_A02_s1_w390BAB393-0635-4160-82E3-5FF0DA618AE1.tif | compound | BRD-A41941932-001-03-0 | 2.312793797 | H-BIOA-002-1 | OC[C@H]1OC(C@HC@@H[C@@H]1O)c2c(O)cc(O)c3c(=O)cc(oc23)c4ccc(O)cc4 | A02 | 20585 | Vitexin | Prestwick Chemical Inc. | 1 | 1.26754E+12 | 20585/ | 20585/ | 20585/ | 20585/ | 20585/ |
おそらく元のメタデータでもCellProfilerで扱えるはずなのですが,自分が初心者で慣れていないため,自分が扱えるようにKNIMEを使って加工しました.
行った処理は単純で,以下に示すように各染色画像ごとのメタデータファイルを作成しました.
この処理によって,5つの染色画像に対応するCSVファイルが出来上がりました.

それではCellProfilerを使ってメタデータを紐付けます.
【CellProfiler】Imagesモジュール
Imagesモジュールは画像を読み込ませるモジュールです.
ここでは先ほどダウンロードした画像ファイルをそのままドラッグするだけです.画像ファイル全部読み込ませると時間がかかるので,一部の画像だけを取り出して読み込ませています.

【CellProfiler】Metadataモジュール
Metadataモジュールの紹介です.
このモジュールでできることの詳細は,また後日行うことにします(複雑だったので…)
今回は行った処理内容を以下に示して,それぞれの簡単な紹介をします.
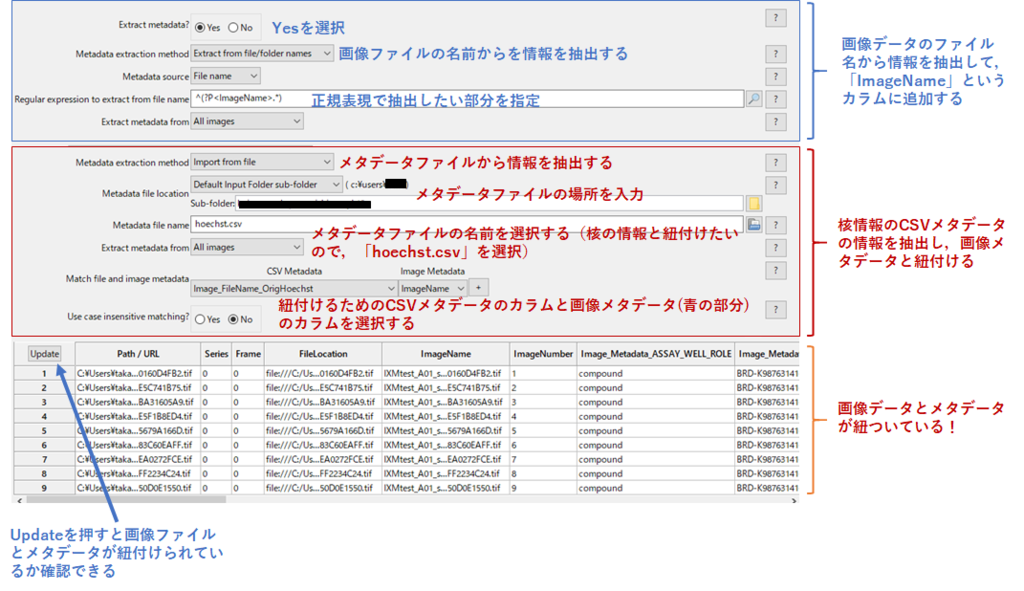
まず,メタデータ処理を行う際は,「Extract Metadata」を「Yes」にします.
続いて,上の青枠の部分ですが,ここでは画像ファイルの名前から情報を抽出しています.
画像ファイルの名前から情報を得るときは,「正規表現」という方法で欲しい部分を選択します.
正規表現 - Wikipedia
正規表現の書き方は以下を参考にしました.
Regular Expressions Cheat Sheet by DaveChild - Download free from Cheatography - Cheatography.com: Cheat Sheets For Every Occasion
今回は,画像ファイルの名前を全て抜き出して,「ImageName」というカラムに格納しました.
続いて,真ん中の赤枠部分ですが,ここではメタデータから情報を抽出します.
ここではCSVファイルが置いてある場所を選択します.核染色画像に対応するメタデータが欲しかったので,hoechst.csvというファイル(KNIMEで加工したファイルです)を選びました.
続いて,「Match file and image metadata」という部分で,メタデータを紐付けるためのCSVメタデータのカラム(Image_FileName_OrigHoechst)と画像メタデータ(ImageName)のカラムを選択します.
同様の操作を,染色画像ごとに行いました.
最後に「Update」ボタンを押すと,きちんと画像ファイルとメタデータが紐付けられていることがわかります!
【CellProfiler】NameAndTypes
ここでは画像データと染色情報を紐付けます.
画像が少ない場合は,一枚ずつ「核染色」,「細胞質染色」…などとできましたが,大量の画像ファイルだと困難です.
そこで,先ほどのメタデータを活用して,画像ファイルと染色条件を紐付けていきます.
NameAndTypesの画面を以下に示しています.
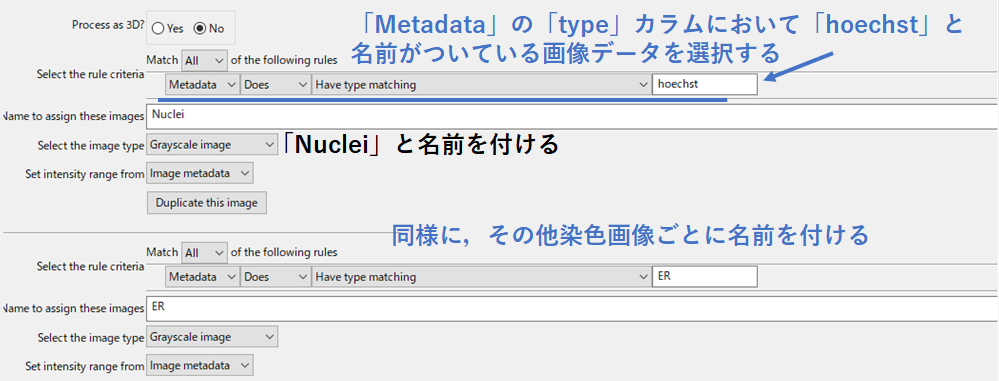
このように,「Metadata」「Dose」「Have type matching」「hoechst」とすると,「Metadata」の「type」カラムで「hoechst」と名前がついている画像データを選択することができます.
ちなみに,青字の「type」は,KNIMEで用意したカラムです.
そして,「Name to assign these images」で「Nuclei」と名前を付けます.これで核染色画像をまとめることができました.
あとは同様の操作を行っていきます.
最後に,メタデータの情報と対応付ける作業を行います. ここでは,プレート番号,Well番号,画像番号(field番号)で対応付けを行いました.
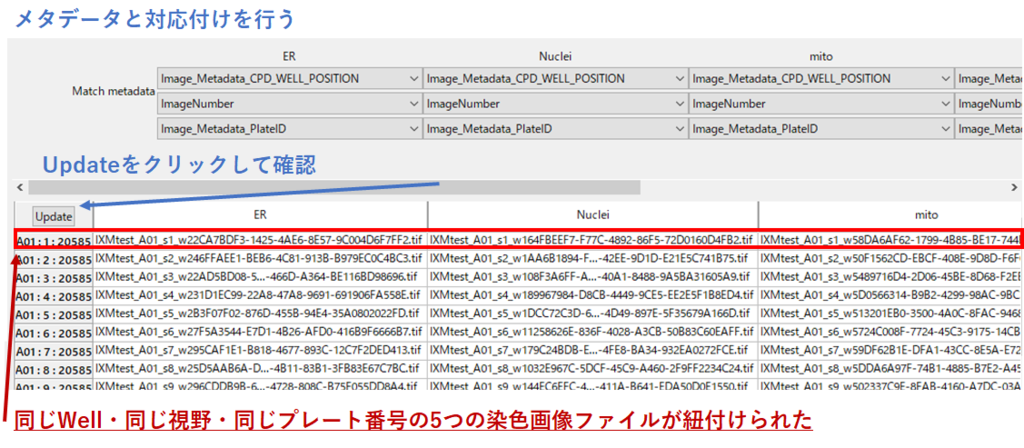
「Update」で確認すると,上図のように,同じWell・同じ視野・同じプレート番号の染色画像を紐付けることができました.
最後に
今回は,画像ファイルとメタデータを組み合わせて名前を付けるところまで行いました.
実は,今まではExample pipelineの紹介だったのですが,ここで初めて実際のデータから画像解析を行うことをしています.
Metadataの部分は,単なる前処理と思っていましたがここもデータを解析する上では大変重要だと感じました…(簡単にできなかったので)
このあと,いよいよ各染色画像ごとに特徴量を抽出して,解析を行いますので,次回以降のブログでこの続きを紹介したいと思います.