こんにちは,@PKです.
創薬スクリーニングで使用されるHCS(high content screening )では,様々な細胞内の特徴量を解析することができます.
【HCS】High Content ScreeningとCellProfilerについて - t_kahi’s blog
その中でも細胞内の粒子(Speckles)を解析したいというニーズは結構あるのかな,と個人的には感じています.
他の評価系では細胞内の粒子を定量的に解析することは難しいですが,HCSを使うことで,非常に簡単に粒子の特徴量を抽出することができます.
今回はCellProfiler内にあるexample pipelinesの「Speckle Counting」を例にして,細胞内の粒子を解析するモジュールについて説明します. 大まかな解析の流れは通常と同じなのですが、粒子解析のために少しだけ前処理が入るので、その点を中心にお話しします.
「Speckle Counting」は以下のサイトからダウントードできます.
CellProfiler | Free open-source software for measuring and analyzing cell images
Speckle Counting pipelineの概要
「Speckle Counting」のPipelineの簡単な説明と,解析フローを図式化したものを以下に示しています.

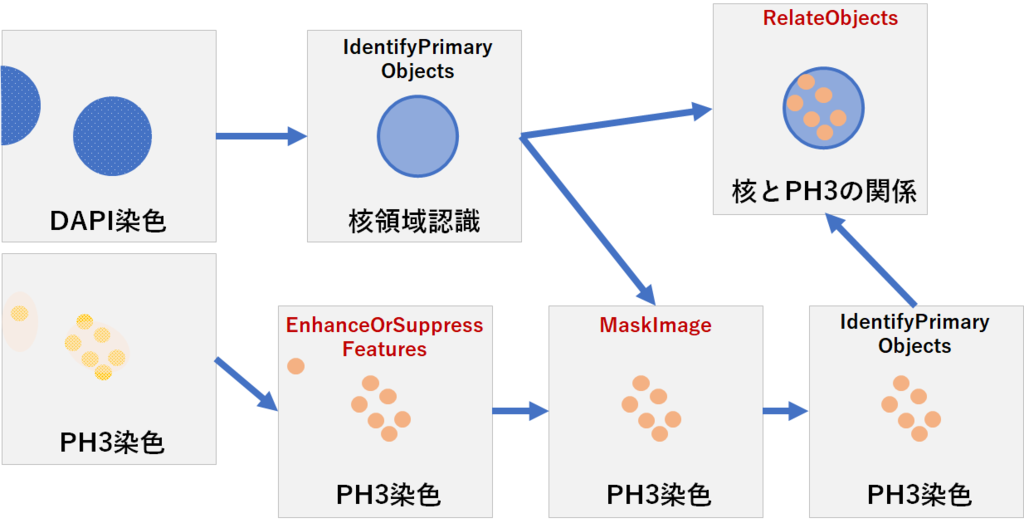
細胞内の粒子を解析するモジュールで特に重要なのが「EnhanceOrSuppressFeatures」と「MaskImage」の部分です. 粒子の画像を解析するときに、粒子の認識精度を上げるため、画像に前処理を施しています.
今回使用する画像について
「Images」モジュールで今回解析する画像を読み込みます.
今回は核染色の画像(左)と,核内のH2AX染色画像(右)の2枚を用いて,画像解析を実施しています.
H2AXの画像は,核内で粒子状の特徴を持つことがわかりますが,若干画像がぼんやりしており不鮮明なように感じられます.

今回はSpeckle Counting のpipelineのなかで,重要なモジュールを取り上げて,それぞれを簡単に紹介していきます.
モジュール紹介
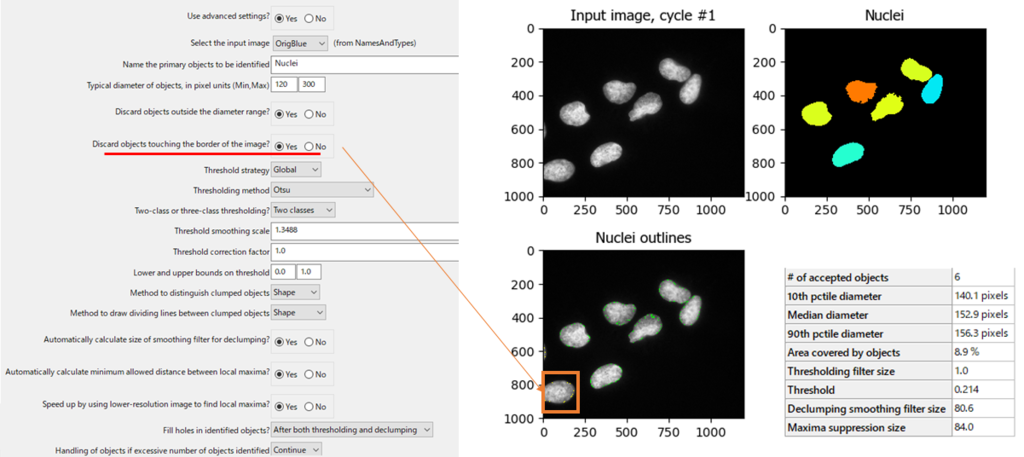
IdentifyPrimaryObjects(Nuclei)
このモジュールでは核の領域認識を行っています.
IdentifyPrimaryObjectsの内容については以前紹介しました.
【CellProfiler】Example pipelineで画像解析の基礎を学ぶ③ - t_kahi’s blog
今回は領域の抽出に大津の二値化を用いており,形状で核を認識しているようです.
このパイプラインの中で重要な点として,
「Discard objects touching the border of the image? 」が選択されていることです.
ここにチェックが入っているので,核領域の認識では一番左下の核はオブジェクトから除外されています.
EnhanceOrSuppressFeatures
今回の粒子解析で一番重要なモジュールです.
このモジュールは画像処理のフィルターを適応することで,画像の特徴(粒子、リング形状、神経突起など)を強調・抑制することができます.
この操作によって,その後のオブジェクトの認識が向上します
モジュールの画面は以下のようになっています.
初登場のモジュールなので,各選択項目についてそれぞれ簡単に紹介します.
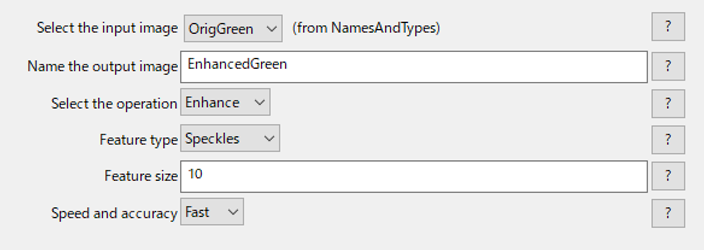
- Select the input image : 強調/抑制したい特徴が含まれている画像を選択する
- Name the output image : フィルターで強調/抑制した画像に名前をつける
- Select the operation : 特徴量を強調/抑制するかを選択する
- Feature type(Enhance) : どのような特徴を強調するかを選択する
- Speckles : white top hat filterを使用して粒子の特徴を強調する.
*参考:Top-hat filterについて
Top-hat transform - Wikipedia - Neurites : 神経突起の強度を高めることができる.「Line structures」または「Tubeness」を選択する
・「Tubeness」 : Image JのTubeness pluginを応用したもの
"Tubeness" ImageJ PlugIn
・「Line structures」 : トップハットフィルターとブラックハットフィルターの差分をとる
*参考:以下の記事が上記フィルター処理についてかなりわかりやすく説明しています.
膨張・収縮・オープニング・クロージング 画像処理ソリューション - Dark holes : Rolling Ball Algorithmを使用してリング状の穴を認識する
*参考:
背景除去について: WAKU-TAKE-A PROGRAM - Circles:circular Hough変換を利用して画像から円の特徴を抽出する
*参考:ハフ変換についてはわかりやすい日本語の記事が多くあり,有難いです.
やさしいハフ(Hough)変換講座 - Qiita - Texture:テクスチャ―(質感)のある特徴を抽出する
- DIC:画像のせん断方向に対して垂直な方向に積分することにより微分干渉顕微鏡画像の光学濃度を回復させる
- Speckles : white top hat filterを使用して粒子の特徴を強調する.
以下に,「EnhanceOrSuppressFeatures」の画像処理前と後の結果を示しています.
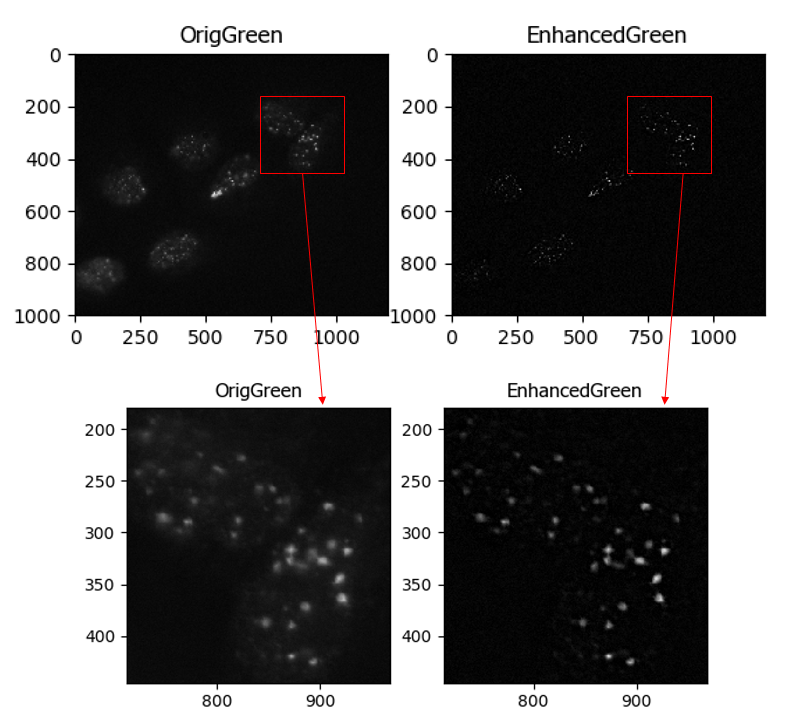 今回はH2AXの粒子画像を鮮明にしたいので,「Enhance」を選択します.
今回はH2AXの粒子画像を鮮明にしたいので,「Enhance」を選択します.
続いて,粒子の特徴を強調したいので,「Speckles」を選択し,Feature sizeに認識したい粒子の最大直径を入力します.
画像処理の結果,若干ぼんやりとしていた粒子がはっきりとしているのが定性的にもわかりますね!!
MaskImage
「MaskImage」はある画像の一部の特徴を別の画像データを使ってマスクすることができるモジュールです.
モジュールの画面は以下のようになっています.
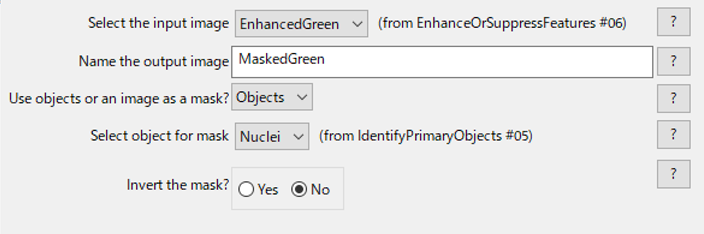
まず,編集したい画像として先ほど粒子を強調した「EnhancedGreen」画像を選択します.
続いて,先ほどIdentifyPrimaryObjectで認識した核オブジェクトを用いてマスクをしたいので,「Objects」&「Nuclei」を選択しました.
その結果を以下に示しています.
左側がオリジナル画像で,右側がマスクされた画像です.

オリジナル画像では左下の赤枠内に粒子がありますが,マスクされた画像では消えています.
これは,IdentifyPrimaryObject(Nuclei)で核オブジェクトを認識したときに,核が画面の端に触れていたので解析から除外されていたので,このような結果になっています.
あるオブジェクト内の粒子のみを認識したいときには,「MaskImage」は便利ですね.
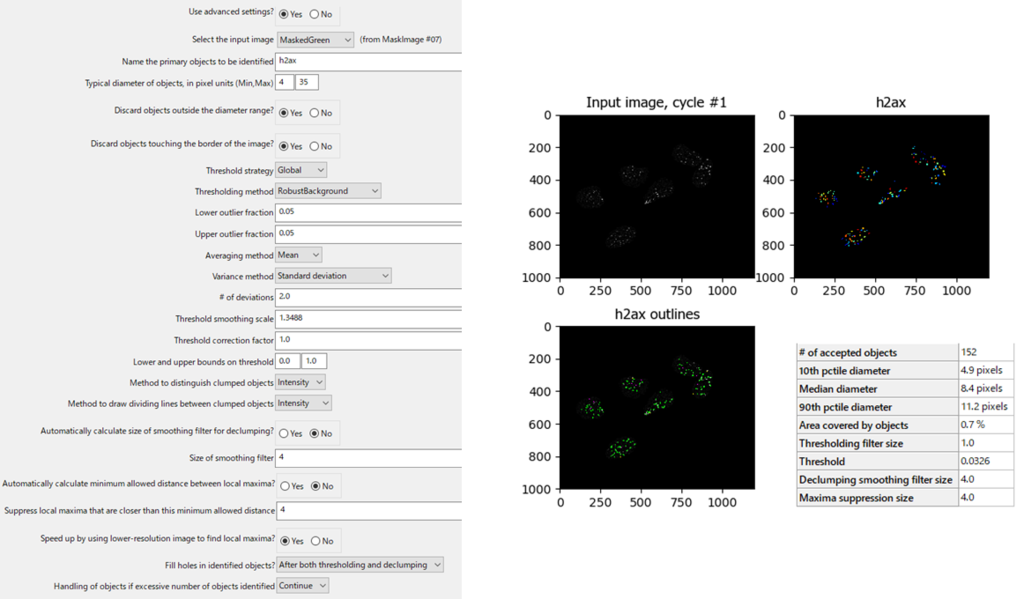
IdentifyPrimaryObjects(H2AX)
ここでは「EnhanceOrSuppressFeatures」と「MaskImage」で強調したH2AXの粒子をオブジェクトとして認識させています.
Thresholding methodはRobustBackground を使用しています.
この手法に関しても,以前の記事を参考にしてください.
【CellProfiler】Example pipelineで画像解析の基礎を学ぶ③ - t_kahi’s blog
画像解析の結果,以下のように粒子オブジェクトがきれいに認識できていることがわかります.
最後に
ここまでで,核とH2AXのオブジェクト認識が完了したので,あとはそれぞれを「RelateObject」で紐づけた後にピクセル強度値を測定して出力すればゴールです!
この部分はCellProfilerの通常の解析の流れなので,説明は割愛しました.
今回example pipelinesの「Speckle Counting」を使って,細胞内の粒子を解析する方法の中でも,粒子の認識モジュールを中心に紹介しました.
様々なHCS関連の論文で細胞内の粒子を測定する評価系を用いています.
今後はそのような論文も紹介していきたいと思います.