前回,MGIのサイトから表現型の全データをダウンロードしてきて,KNIMEを使ってまとめてみました.
http://t-kahi.hatenablog.com/entry/2018/05/01/003455
全データを取得することができるのですが,ある特定の表現型のデータだけで十分という場合は多いと思います.
そこで,今回はMouseMineというサイトから任意の表現型のデータをとってきてKNIMEを使ってまとめてみました.
MouseMineのサイトへ飛んで,中央右下のPHENOTYPEのバーを選択し,Mammalian phenotypes (MP terms) ⇒ Mouse genes and modelsをクリックすると
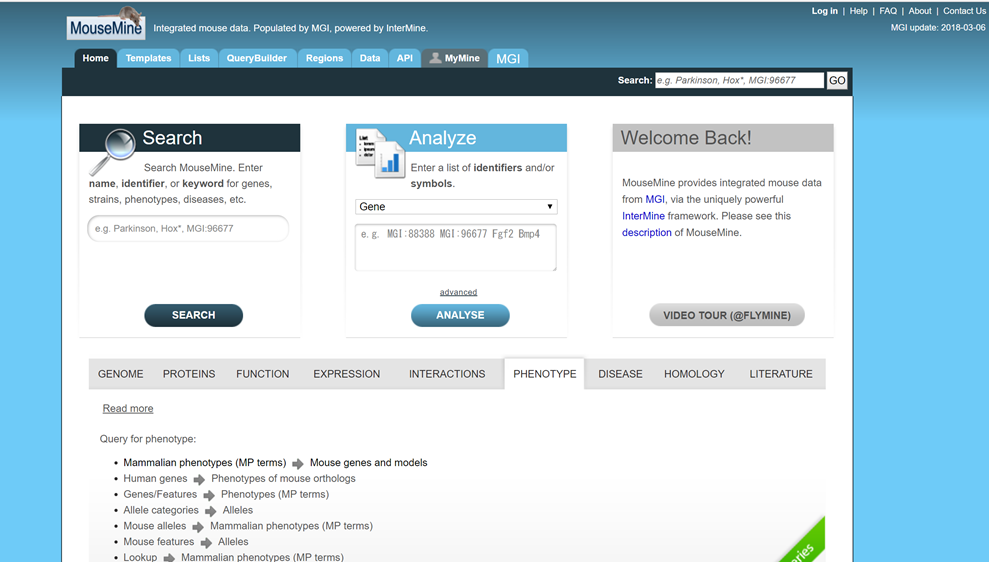
以下のようにMP termを打ち込むと表現型をまとめて取得できるページへ移る(便利…!!)
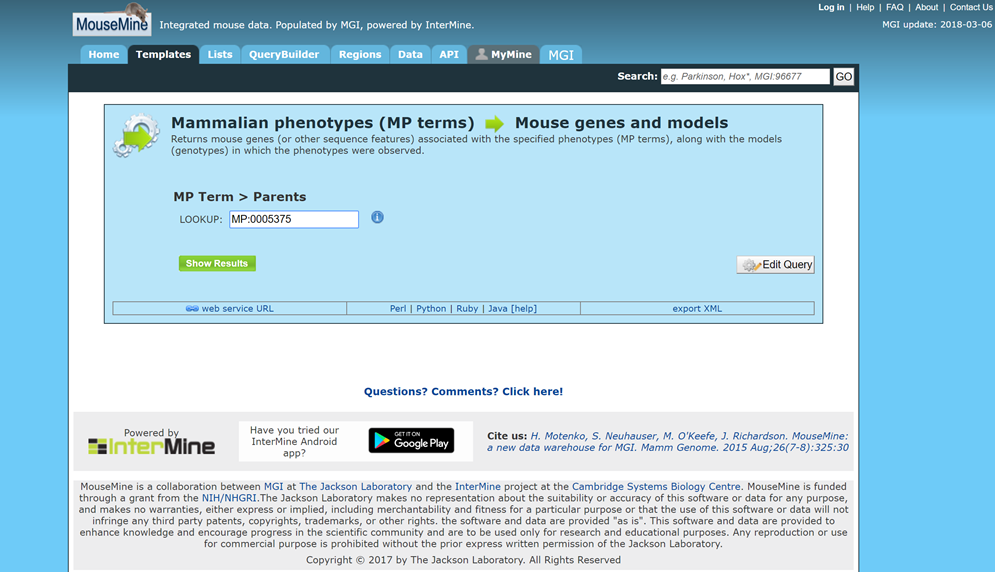
MP termはMGIのサイトのMammalian Phenotype Browserから自分の好きな表現型とそのMP termを検索することができます.
下の画像だと,adipose tissue phenotypeはMP:0005375ということがわかります.

これを先ほどのMouseMineの検索窓に打ち込むと,以下のようにadipose tissue phenotypeに関連した表現型情報がザーッと出てきます. さらに,この検索ではadipose tissue phenotypeの下流のツリーすべての情報をとることができる.
画面右のExportのタブから,csv fileを出力して自分のローカルフォルダに入れておきます.

ここで得た情報だけでは少し使いにくいので簡単にKNIMEを使ってまとめます. 例として,「lethality」を含む表現型を示す遺伝子を抜き出すことを行います.
下の画像から,mortality/aging⇒abnormal survivalにその情報が入っていることがわかります.
しかし,abnormal survivalのツリーの下には「extended lifespan」という表現型が含まれており,これは「persistence of life for a longer period than is normal for an organism」ということから今回はabnormal survivalからextended lifespanを除いたデータを作成します.

KNIMEのフローはこんな感じです

ポイントを説明をすると
- Node1のCSV readerでabnormal survivalの情報を読み込む
- Node2のCSV readerでextended lifespanの情報を読み込む
- Node3のReference Row Filterでextended lifespanを除く
ここまでで,望みの表現型のデータを得ることができましたが,データを見てみると,遺伝子名はMice symbolで表されているので,このままほかのデータと組み合わせたりすることが少し難しい..

そこで,homologene IDを使ってhuman symbolに変換しておきます.
homologene IDは種間の相同的な遺伝子を紐づけることができます.
miceとhumanのhomologene IDのリストはMGIのサイトから手に入れることができるので,これをcsv fileでダウンロードしておきます.
もう一度KNIMEのワークフローに戻り,Node10のCSV readerで先ほどのhomologene IDリストを読み込みます.
ファイルを見てみると下リストのように,humanとmiceの遺伝子がhomologene IDを使って紐づけられていることがわかります.

- Node11のRole based row splitterでhuman Taxon IDを参考にして,humanとmiceのデータをそれぞれ別々に分離しておきます.

- Node12 Joinerで先ほどの表現型データと,mice symbol +homologene IDのデータをつなげると,下リストのように表現型データにhomologene IDをつなげることができます.

- Node16 joinerでmice symbol +homologene IDと結合させることで,先ほどの表現型データとヒトのsymbolやEntrez gene IDを紐付けることができた.

もちろん,単純に表現型データを取ってくるだけでも良いのですが,得られたデータを使うことを考えると,紐づけがしやすいタグを付けておくほうが良いかと感じました.