創薬ターゲット分子の重要な情報の一つに,ノックアウトマウスの表現型データがあります.
例えば,何か標的分子の阻害剤を作りたいと考えたとき,表現型のデータを見ることで,その遺伝子を抑えた際に一番強く出る作用を観察することができます.(もちろん,発生の段階で重要な因子であれば胎生致死などの表現型になってしまい,欲しい情報を得ることができませんが)
私はよく,MGIというサイトを使って,マウスの表現型データを調べます.
MGIの説明はintegbioデータベースカタログのサイトで紹介されています.
MGIとはマウス研究の総合サイトです。マウス遺伝子に関する情報の他、遺伝子発現データ、多型・変異情報、パスウエイ、遺伝性癌症候群モデルラットに関するデータ、マウス各系統の表現型に関する詳細な情報等を提供しています。
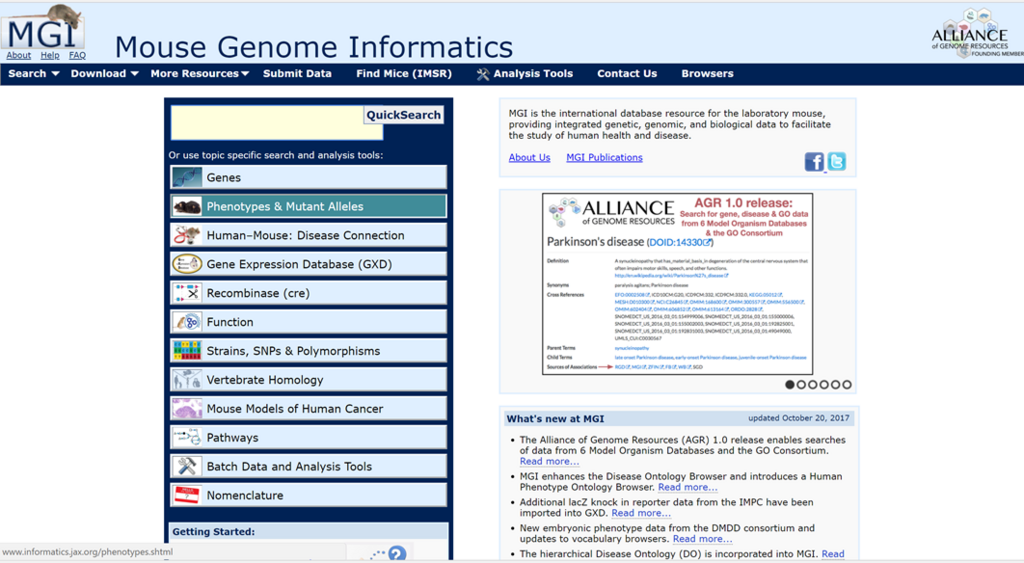
MGIのサイトへ飛ぶと,下記のような画面がでてきます.
このなかの,phenotype & mutant allelesから,様々な表現型の情報を得ることができます.
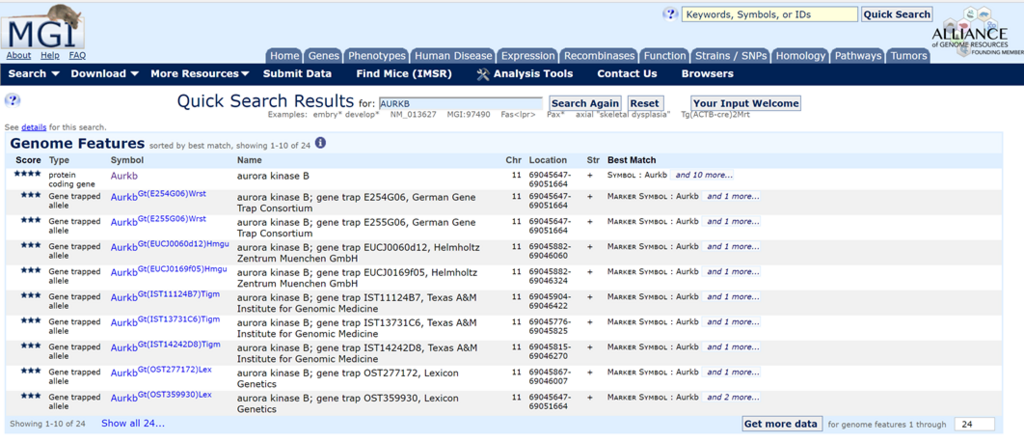
例として,Aurora-Bという遺伝子(gene symbol : AURKB)を調べてみます.
QuickSearchでAURKBで検索をすると,トップに遺伝子が出てきます.
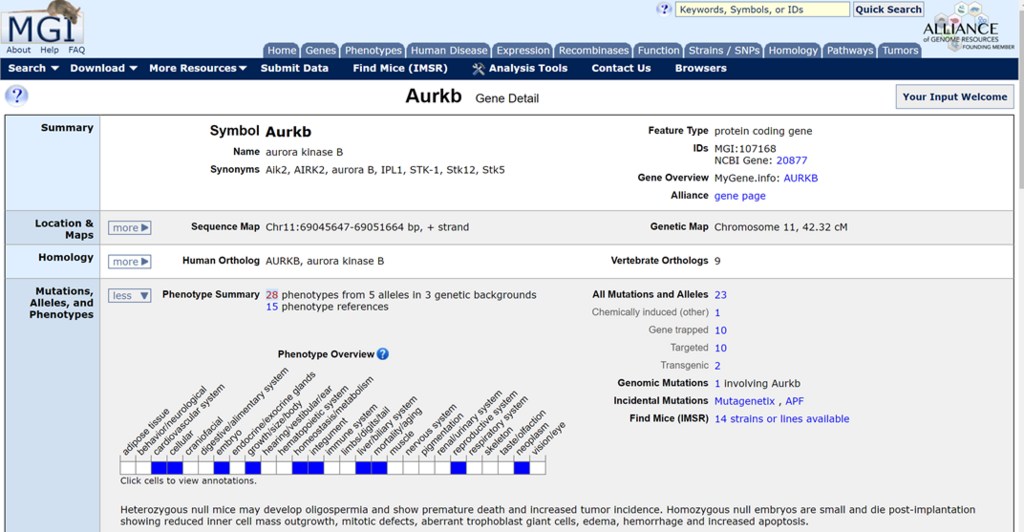
ここでは,Aurora-Bに関連した様々な情報を見ることができますが,今回はphenptype summaryのボタンをクリックして,表現型を確認します.
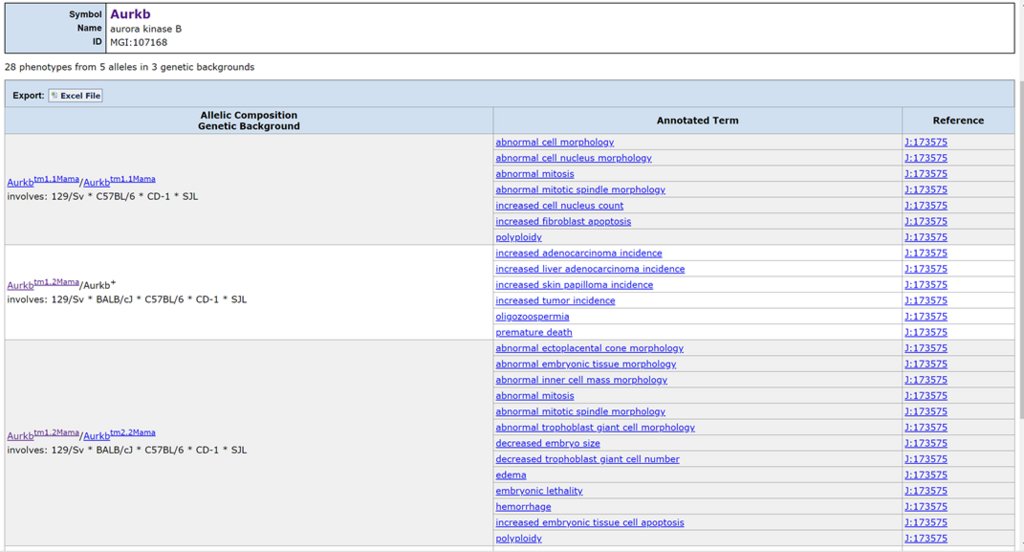
すると,以下のように,Allelic Composition, Genetic Backgroundに対応した表現型がでてきます.
それぞれの表現型のさらに細かい情報も見ることができます.
ここまでは,マウスの表現型情報を入手するときに行っていたやりかたですが,最近ターゲット分子をまとめて調べることが多く,この調べ物を10回以上繰り返してポチポチみることが億劫になってきたので何とかして簡単にマウスの表現型情報を手に入れられないかと調べていました.
いろんな方にアドバイスしてもらったところ,どうもMGIのデータは全部ダウンロードできるらしいということがわかりました(全然知らなかった…)
やり方は,MGIトップページの左上,DOWNLOAD⇒All MGI Reportsへ移動します.

さらに,Alleles and phenotypesをクリックして,データをダウンロードすることができました.

ダウンロードしたファイルは,このようにタブ区切りの文字の羅列なのでこのままでは扱いずらいので,こちらを少し工夫して見やすくすることを考えます.
http://www.informatics.jax.org/downloads/reports/VOC_MammalianPhenotype.rpt

使用したのは以下の4つのファイルです.
- Mouse/Human Orthology with Phenotype Annotations (tab-delimited)
- human,miceのsymbol,Entrez Gene ID とMGI Allele Accession IDが紐づいているデータ
- List of All Mouse Phenotypic Alleles (tab-delimited)
- MGI Allele Accession IDと Allelic Composition, Genetic Backgroundなどが紐づいているデータ
- All Genotypes and Mammalian Phenotype Annotations (tab-delimited)
- MGI Allele Accession IDと詳細な表現型データが紐づいているデータ
- VOC_MammalianPhenotype
- 表現型とmammalian phenotype ID (MP)を紐づけているデータ
この4つのデータをKNIMEを使ってまとめることにした.
やっていることは非常に単純で,それぞれのデータを紐づけているIDでつなぎ合わせて,一つのcsv fileを作成するだけです.
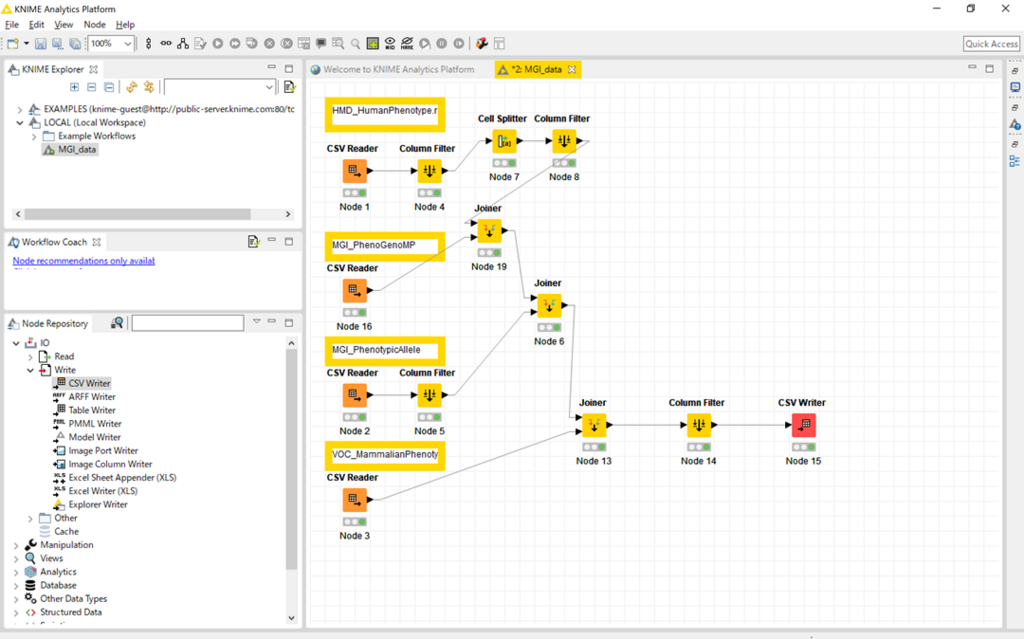
ざっくり説明をすると,
- HMD_HumanPhenotypeをcsv readerで読み込み、使わないカラムを除去 (ヒトの遺伝子名とMGI accession IDを紐付けておく)
- MGI_PhenoGenoMPのデータをcsvで読み込み、MGI accession IDでinner join (それぞれの遺伝子に対して詳細なmammalian phenotypeのデータが入っている)
- MGI_PhenotypicAlleleのデータをcsvで読み込み、こちらはAllele symbolでjoin (Allelic Composition, Genetic Backgroundなどの情報を追加)
- VOC_MammalianPhenotypeがMPと表現型の対応表なので、これをcsv reader で読み込み、先ほどのデータとjoinさせる(ここのjoinはleft outer joinにしておく!)
あとはcsv fileに出力して、それぞれの遺伝子とmice phenotypeの対応表を手に入れることができました.
先ほどと同様に,AURKBで検索をかけてみると,一応それっぽいデータを得ることができている気がする(たぶん…).
手元にデータがあるので,今迄みたいにブラウザを立ち上げて検索窓で一つずつ調べなくても良いし,まとめて自分のPCで処理ができるのは良いなあと思いました.

なんか,抜けや漏れがあると思うので,また追記をするつもりです. データベースからデータを取ってくるとか,KNIMEの使い方は本当に素人なのでとても時間がかかってしまいました..(楽しかったけど)