こんばんは,@PKです.
バイオ実験者にとってはt検定やカイ二乗検定は馴染みがあると思いますが,KNIMEでも統計に関する様々なノードを使って簡単な統計解析を実施することができます.
例えば実験結果のワークフローを作って実験結果の解析をするときに,これらのノードを挟むことでお手軽に検定を行うことができます.
また,カイ二乗検定のようにKNIME上ではノードが無い場合でも,「R snippet」ノードを使うことで検定を行うことができます.
今回は,KNIME上で1標本・2標本t検定を行うノードと,カイ二乗検定について紹介したいと思います.
- KNIME Workflow
- Single sample t-test ノードで1標本のt検定
- Independent groups t-test ノードで対応のない2標本t検定
- R snippetノードでカイ二乗検定
- まとめ
KNIME Workflow
今回は「1標本のt検定」,「対応のない2標本t検定」,「カイ二乗検定」について紹介したいと思います.
それぞれの検定で用いたサンプルデータについては,こちらのサイトで使用されている例を用いております.
データ解析・マイニングとR言語
また,検定については,専用のノードと,「R snippet」で処理した場合の両方を示していきます.
このような形で紹介するのは,KNIMEのノードは応用性が高いわけではないので,もし細かい設定をしたい場合は「R snippet」でRのコードを書いたほうが良いからです.
Single sample t-test ノードで1標本のt検定
まずは1標本のt検定を行います.
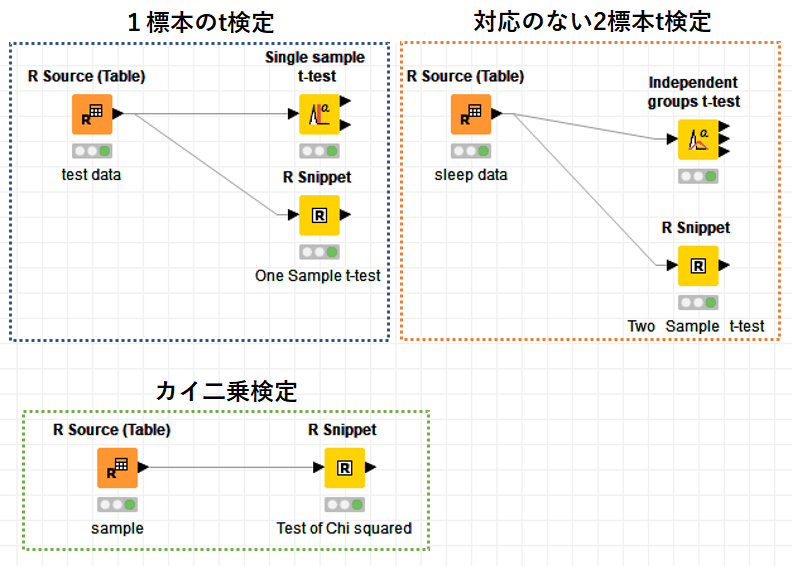
用いるデータは「R Source」で取得しています.
全国平均身長が170.2cm,25人に聞き取り調査を行った結果の身長が示されています.
これらのデータの比較を行います.
#R source x<- c(171.6, 173.6, 167.6, 169.1, 183.0, 173.7, 168.3, 169.9, 182.3, 166.0, 172.8, 184.1, 158.9, 168.1, 168.5, 175.3, 179.6, 170.7, 173.1, 173.6, 169.1, 167.9, 177.8, 171.8, 178.1) data <- data.frame(x) knime.out <- data
KNIMEには「Single sample t-test 」ノードが用意されているので,こちらを使用すればOKです.
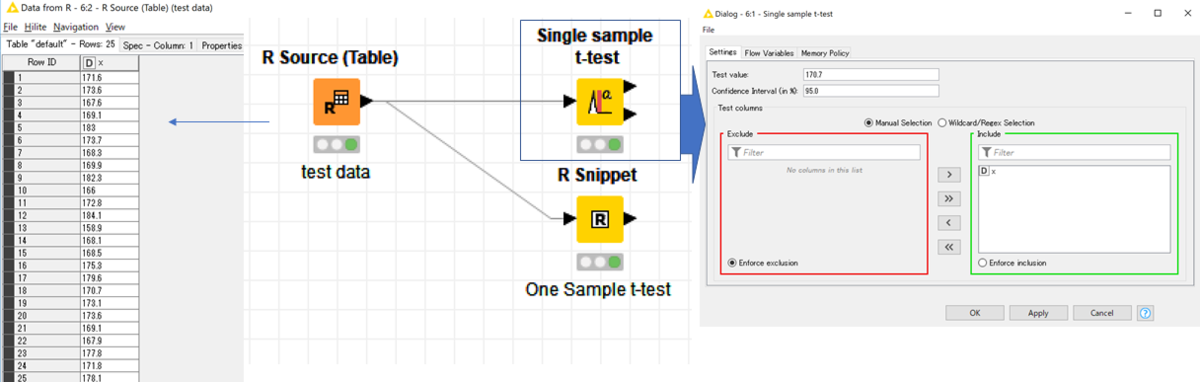
「Single sample t-test 」の設定画面で,比較するカラム(test column)と,数値(test value)と信頼区間を設定します .
あとは実行するだけで結果が返ってきます.
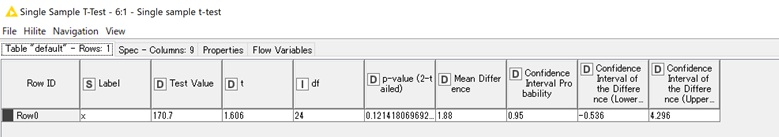
t検定の結果はノードの上側ポートから以下のように出力されます.(下側ポートはtest columnの統計値が出力されます)
同じ事はR snippetノードを使っても行うことができます.
#R snippet #t.test(x, y = NULL, alternative = c("two.sided", "less", "greater"), mu = 0, paired = FALSE, var.equal = FALSE, conf.level = 0.95, ...) t_test <- t.test(knime.in,mu=170.7,alternative ="two.sided") results <- cbind(t_test$statistic,t_test$parameter,t_test$p.value,t_test$conf.int[1],t_test$conf.int[2],t_test$estimate,t_test$null.value,t_test$stderr,t_test$alternative) colnames(results)<-c("t","df","p.value","conf.level_lower","conf.level_upper","estimate","mean","stderr","alternative") knime.out <- results
実行すると以下のように出力されます.

「Single sample t-test 」ノードの結果と一致していることがわかります.
Independent groups t-test ノードで対応のない2標本t検定
続いては対応のない2標本のt検定を行います.
24-3. 2標本t検定とは | 統計学の時間 | 統計WEB
sleepというRのサンプルデータを読み込んで使用します.
Student’s Sleep Data 学生の睡眠データ Data which show the effect of two soporific drugs (increase in hours of sleep compared to control) on 10 patients. 10人の患者の2つの催眠薬(対照と比較して睡眠時間の増加)の効果を示すデータ。
R言語 サンプルデータ一覧 | トライフィールズ
R Sourceでsleepデータを読み込みます.
#R source sleep knime.out <- sleep
対応のないt検定を実施したいので,「Independent groups t-test」ノードを使います.
nodepit.com
*対応のあるt検定のノードもあります.
Paired t-test — NodePit
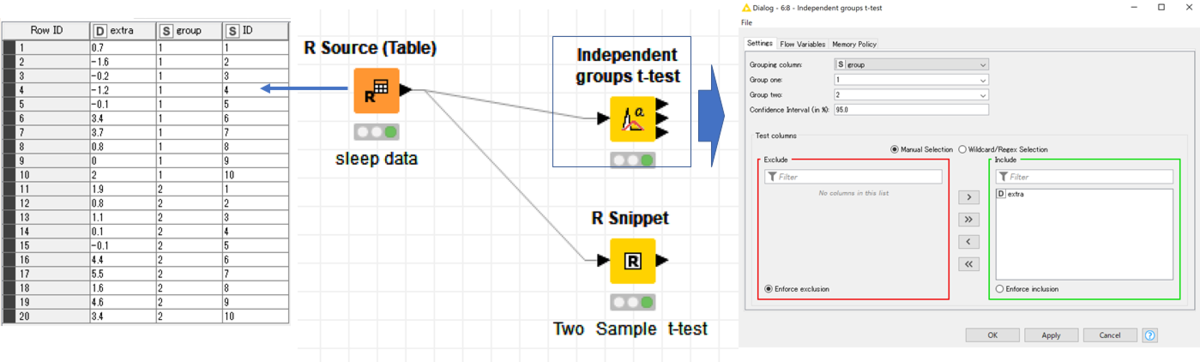
group1とgroup2で睡眠時間の増加(extra)に差があるかを検定します.
「Independent groups t-test」ノードの使い方は簡単で,グループのカラムを選んで,比較したいグループ(group1 & group2)と信頼区間(95%)を選択します.
続いて,テストするカラムを選択して実行します.
t検定の結果を確認する前に,2標本の等分散について検定の結果を確認しましょう.
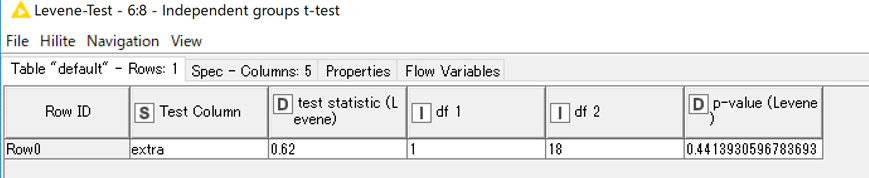
真ん中のポートから等分散性に関する情報として,「Levene-Testの統計値」が出力されます.
等分散の検定はF検定なのかな,と勝手に思っていたので,ここは驚きでした.
Levene-Test知らなかったので,勉強になりました.
ルビーン検定 - Wikipedia
結果を確認すると,p 値は0.62で有意水準0.05より大きいので、両グループの母分散は同じという仮説が採択されます.
t-testの統計値は「Independent groups t-test」ノードの一番上のポートから出力されます.
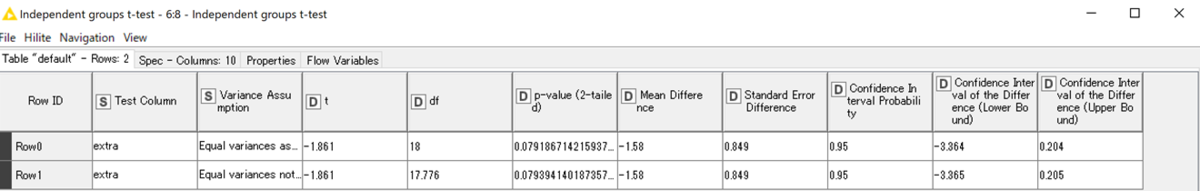
2行結果が出力されていますが,これは等分散・不等分散の両方の結果が示されています.
Levene-Testの結果から今回は1行目の結果を確認します.
p = 0.079ということで両グループ間で睡眠延長に差が無いという結果になりました.
参考として,同じことを「R snippet」ノードで表す際のコードを示します.
#R snippet #var.test(x, y, ratio = 1, alternative =c("two.sided", "less", "greater"), conf.level = 0.95, ...) v_test <- var.test(extra~group,data=knime.in) a <- v_test$p.value t_test <- t.test(extra~group,var.equal = TRUE,data=knime.in) b <- t_test$statistic c <- t_test$parameter d <- t_test$p.value e <- t_test$conf.int[1] f <- t_test$conf.int[2] g <- t_test$estimate[1] h <- t_test$estimate[2] results <- data.frame(a,b,c,d,e,f,g,h) names(results) <- c("var.test_p.value","t","df","p.value","confidence_interval(lower)","confidence_interval(upper)","group1_mean","group2_mean") results knime.out <- results
上のコードでは,F検定を挟んで等分散性を確認してから等分散のt検定を実施しています.
以下出力結果より,「Independent groups t-test」ノードと同じ結果が返ってきていることがわかります.

R snippetノードでカイ二乗検定
下のサイトでも言われているように,KNIMEにはカイ二乗検定ができるノードはありません…
Test of Chi squared - KNIME Analytics Platform - KNIME Community Forum
なので,カイ二乗検定をKNIME上で行いたい場合は「R snippet」ノードを使いましょう.
R Sourceでサンプルデータを入力します.
#R source a <-matrix(c(101,120,70,35,153,162,88,46,89,135,78,24),3,4,byrow=T) knime.out <- a >a [,1] [,2] [,3] [,4] [1,] 101 120 70 35 [2,] 153 162 88 46 [3,] 89 135 78 24
各行がグループを表しており(1~3グループ),各列が質問に対する回答者数を表しています(質問1~4). この質問に対する回答者数の傾向がグループ間で異なるかどうかを解析します.
「R snippet」ノードでデータを出力する際のコードを示します.
#R snippet chi_test <- chisq.test(knime.in) a <- chi_test$statistic b <- chi_test$parameter c <- chi_test$p.value results <- data.frame(a,b,c) names(results) <- c("X-squared", "df", "p.value") knime.out <- results
結果は下図の通りで,p = 0.214ということなので,質問に対する回答者数の傾向は各グループで同じということが言えます.

まとめ
統計の基礎的な部分ですが,t検定に関するノードの紹介と,Rノードを使ったカイ二乗検定について紹介しました.
個人的には「R snippet」で処理を行った後にその結果をテーブルとしてまとめて次のノードへ渡す部分は少し面倒かも,と感じました.
ただ,カイ二乗検定のようにRでしたできない処理もあるので,「R snippet」ノードを使えるようになっておいて損はないと思います.