約3か月ぶりの投稿になってしまいましたが,久々にKNIMEに関する記事を書いています.
今日は,96プレート,384プレートなどを使って,酵素アッセイや細胞評価アッセイを行った結果から,シグモイドのフィッティングやIC50値の算出を行う,ということをKNIMEを使って示したいと思います.
評価するプレートレイアウトと,生データ(おそらくcsvやexcelのテーブル形式のデータ)からコピペをせずに直接アッセイの解析結果が出てきます.
自分は業務上,微妙に条件をいじりながら細胞アッセイを96プレートや384プレートで行っているので,この辺を自動化できると大変役に立ちます.
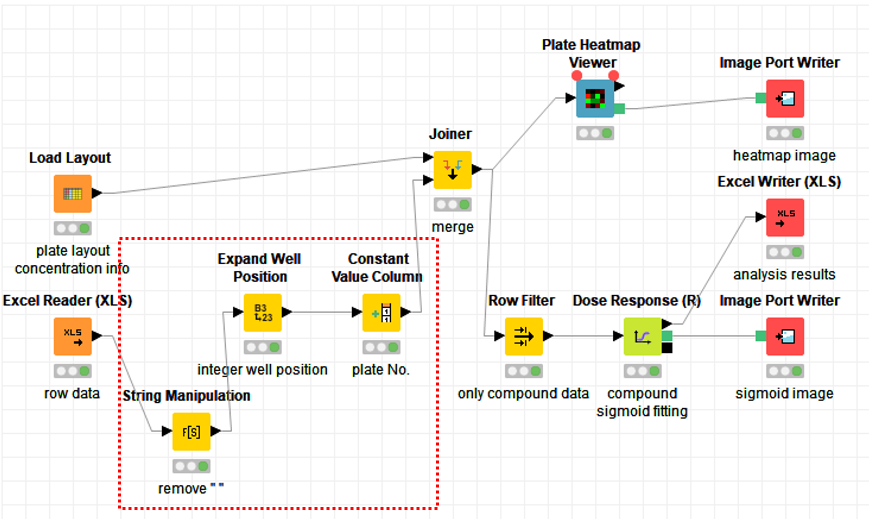
今回のKNIMEのフローは以下の通りです.
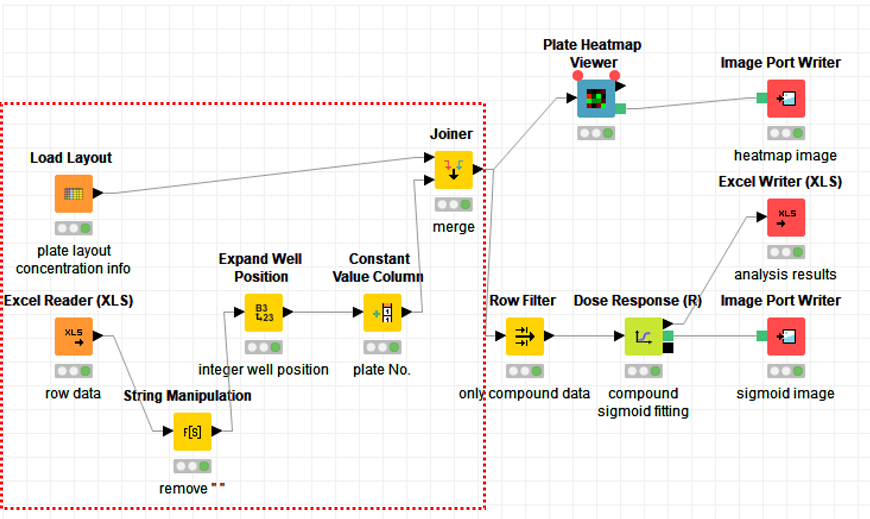
この記事では,前半部分のアッセイを行ったプレートのレイアウト情報と,アッセイの結果得られた生データを紐付けする部分(赤点線囲い)について説明をします.
KNIMEに限りませんが(spotfireとか),テーブルデータ以外の,プレート配置のようなデータを読み込むことは,少し特殊な操作が必要です.
KNIMEでは,Excelファイルを読み込むExcel Readerというノード以外に,プレートレイアウトを読み込むことができるLoad Layoutというノードがあります.
Load Layoutは,community nodes⇒ HCS tools⇒Utilitiesにあります.
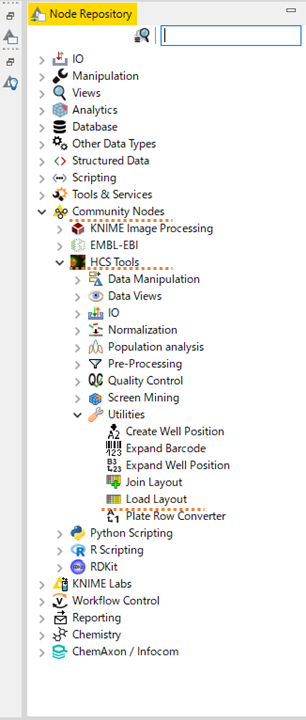
HCS toolsの詳細は以下のサイトを参照してください.
High Content Screening Tools (trusted extension) | KNIME
https://github.com/knime-mpicbg/HCS-Tools/wiki
Load Layoutを開いてみると読み込むファイルを選択するよう指示がありますが,どのように書けばよいのかわかりせんでした...
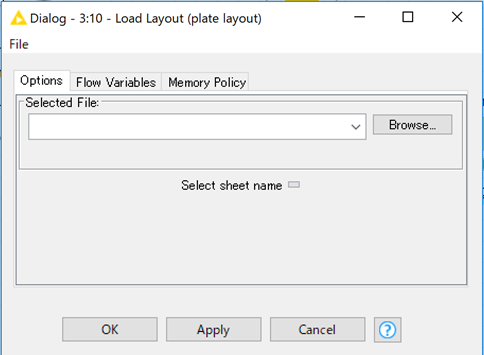
そんな時はdialog画面の右下の?マークをクリックすると,ノードの説明画面が出てきます.
The node is dedicated to ease the processing of microscope or plate reader data. It loades meta information and/or layout data from a sheet of an Excel file which follows a specific formatting. The sheet can contain different layers of information (e.g. compound name, compound concentration, ...).
The first layer has to start at C5 in the Excel sheet. This position contains the layer name (used as column names in the output table). The next row (starting at D6) contains the plate column numbers (1,2,3,...), while column 5 (starting at C7) contains the plate row identifier (A,B,C,... or 1,2,3,...). The dimensions are limited to 1536 well format (32 rows, 48 columns). Dimensions have to be the same for all layers. Within the area defined by plate row and column identifiers cells can contain metainformation of different data types (string, double, integer) or left empty. The next layer starts again with its name in column C with a gap of 2 rows to the previous layer. Each layer will result in a new column of the output table.
まとめると
- C5にプレートの名前
- D6からcolumn numberを記載する
- C7からrow numberを記載する
- 続けてプレートを加える場合は2行空ける
ということらしいので,エクセルでプレートレイアウトを作りました. 情報としては
- wellの配置情報(ブランク,min, max, ポジコン,化合物など)
- 化合物の濃度情報(IC50が欲しいので)
を記載しました.
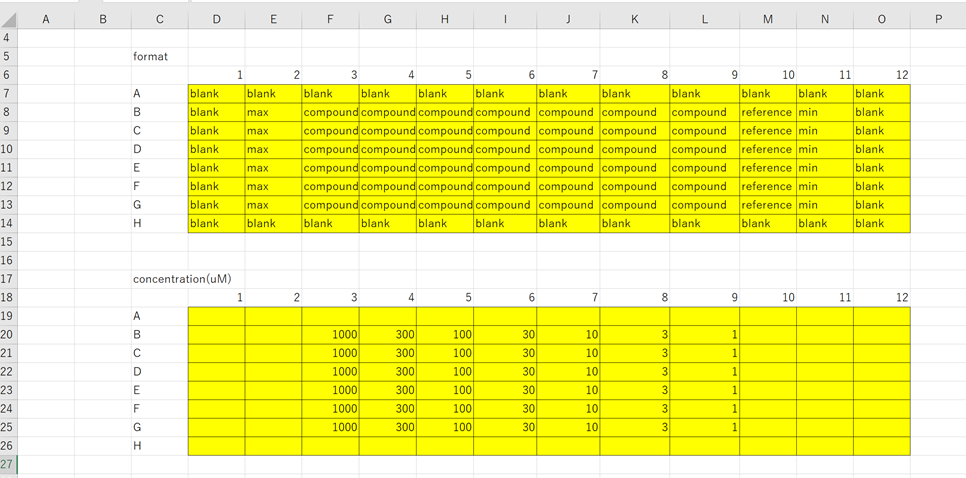
上記エクセルファイルを,Load Layoutで読み込ませると,以下のようなデータが出てきました.
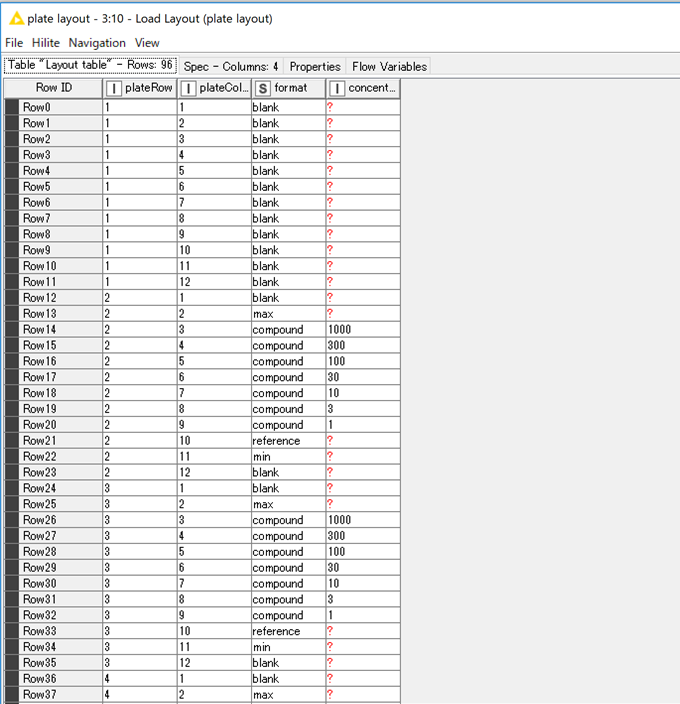
plate RowはA,B,C...だったのが1,2,3...に変換されており,対応するRowとColumnの部分にwell配置と,濃度(化合物)が記載されたテーブルが出来上がりました.
よくあるエクセルのプレート形式のフォーマットがこんなに簡単にテーブル形式で出てくるのはちょっと感動しました.
さて,解析データに関しては,Excel readerで読み込むだけなので詳細は割愛します.ほしいデータの部分だけ抜き出して,以下のように読み込ませました. Col0が,wellとcolumnの番号,Col1がアッセイデータになります.
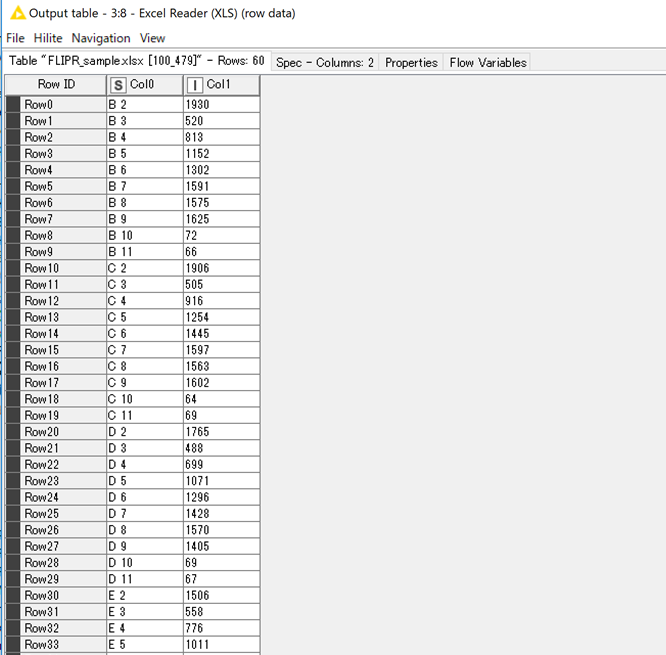
ただし,このままだと対応するカラムがないため,joinerでつなぐことはできませんので,Col0の列を少し修正する必要があります.
その操作は以下の赤点線囲いの部分で行います.
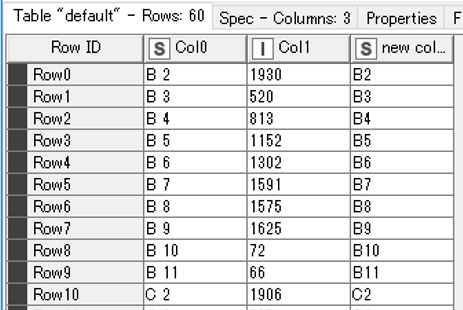
まずはstring manipulationでCol0の「B 2」の間のスペースを除きます.
(なぜこんなことをするかというと,次のExpand Well Positionの操作で,スペースが邪魔だからです)

スペースを除いたnew columnが生成されていることがわかります.
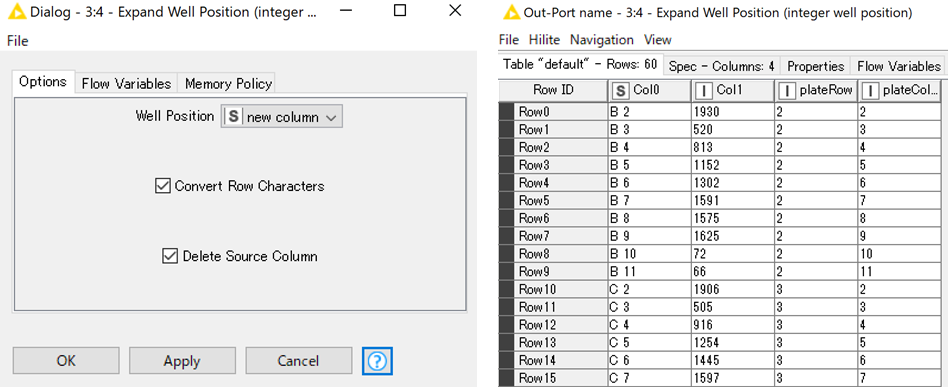
続いて,Expand Well Positionで,B2, C5のようなwellポジションを2つのカラム(row number, column number )に変換してくれます.
このノードも,community nodes⇒ HCS tools⇒Utilitiesにあります.
以下は,Expand Well Positionのnode descriptionです.
This node takes a string column containing well positions (like B3 or C14) and appends two integer columns named 'plateRow' and 'plateColumn' containing two integer coordinates of the wells (link 2 and 3 or 3 and 14). It handles well positions up to 1536 plate format.
Additional splitting characters (comma, colons, semicolons, underscore) between the row letter and the column number will be ignored.
この説明とおり,「B3」のようにアルファベットと数字の間は空いていないことが前提なので,先ほどの処理を行いました.
Expand Well Positionでは,well positionのカラムを選ぶだけで,下の右の結果のようにplate rowとplate columnを分けることができました.
このノードを使わなくてもstring manipulationとかを使うとバラバラにできるのですが,専用のノードがあると便利ですね…
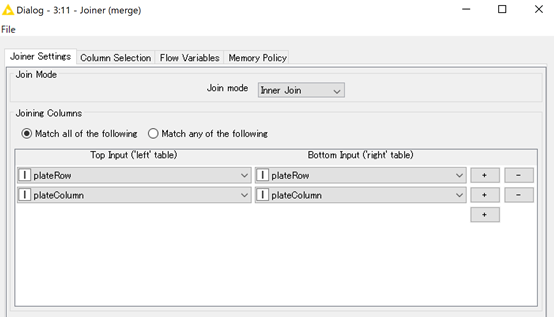
最後に,constant value columnでplateの名前を入れ(プレートが複数ある時は名前を付けたほうが便利かと思います),joinerでつなぎます.
プレートレイアウトも,生データもlateRowとPlateColumnという共通するキーを持っているので,以下のようにつなげます.
Joinerの処理を行うと,プレート配置と生データの結果がそれぞれマージされたことがわかります!

一度フローを作ってしまえばあとは使いまわしできるので,同じようなデータ形式なら簡単に同じことができますね.
この後のheatmapの表示や,シグモイドのフィッティングなどは次の記事で紹介したいと思います.
皆さんはこの辺の作業をどのように行っているか(コピペ,excelマクロ,python,spotfireなどなど)興味があるので,ぜひ教えていただけると幸いです.