こんばんは,@PKです.
ある遺伝子のリストに含まれる遺伝子をシグナルや機能ごとにクラスタリングをしたいと思い,調べていたところ,Dr.Bonoの本にMetascapeを使って遺伝子リストから機能の情報を取得する項目がありました.
今更なのですが,このDr.Bono本は非プログラマのバイオ系が混乱するインフォマティクス解析の事柄に対して細やかなTipsを散りばめているので本当に勉強になります.
今回は,遺伝子データセットをシグナルや機能ごとにクラスタリングすることについて紹介したいと思います.
さて,Metascapeにアクセスすると以下のような画面に飛びます.

自身の持っている遺伝子のリストをアップロードするだけで,簡単に解析を行うことができます.
Metascapeの使い方はこちらを見て勉強させてもらいました.
togotv.dbcls.jp
今回は,以下に示すMetascape single listのサンプル遺伝子リストを使い,Metascapeでシグナルに関する情報を取得してRで階層型クラスタリングとヒートマップを出力しました.
| Gene | OptionalDataColumns |
|---|---|
| UBL5 | 2.6 |
| NDUFB8 | 3.5 |
| CHMP2B | 4.9 |
| PRPF8 | 2.9 |
| NOSTRIN | 4.2 |
| MFAP1 | 3.7 |
| CWC22 | 2.1 |
| PLCH2 | 3.1 |
| PRPF31 | 3.9 |
| ATP6AP1 | 3 |
| ... | ... |
Rでのヒートマップは以下を参考にしました.
heatmap.2 function | R Documentation
ヒートマップ | R で遺伝子発現量などをヒートマップに描く方法
コードを以下に示します.
library(gplots) library(openxlsx) library(dplyr) read <- read.xlsx("D:/metascape/metascape_result.xlsx") data <- data.frame(read) #check columns name names(data) #remove unused columns data <- data[,c(1,17:31)] #check data type and change character to number str(data) data %>% mutate_at(vars(-Gene), as.numeric) -> data_2 str(data_2) #change rowname as gene name rownames(data_2) <- data_2$Gene data_2 <- data_2[c(2:16)] names(data_2) #calculate distance & clustering d1 <- dist(data_2, method="euclidean") d2 <- dist(t(data_2), method="euclidean") c1 <- hclust(d1, method="ward.D") c2 <- hclust(d2, method="ward.D") #export heatmap data png("data.png", width=3600, height=3600) plot <- heatmap.2(as.matrix(data_2), col=colorpanel(256, low = "lightblue", high= "tomato"), Rowv=as.dendrogram(c1), Colv=as.dendrogram(c2), cexRow=4, cexCol=4, margins=c(70,20), density.info="none", trace ="none", key ="FALSE") dev.off()
細かいのですが,縦軸が遺伝子,横軸がそれぞれのシグナル(機能)を示しています.
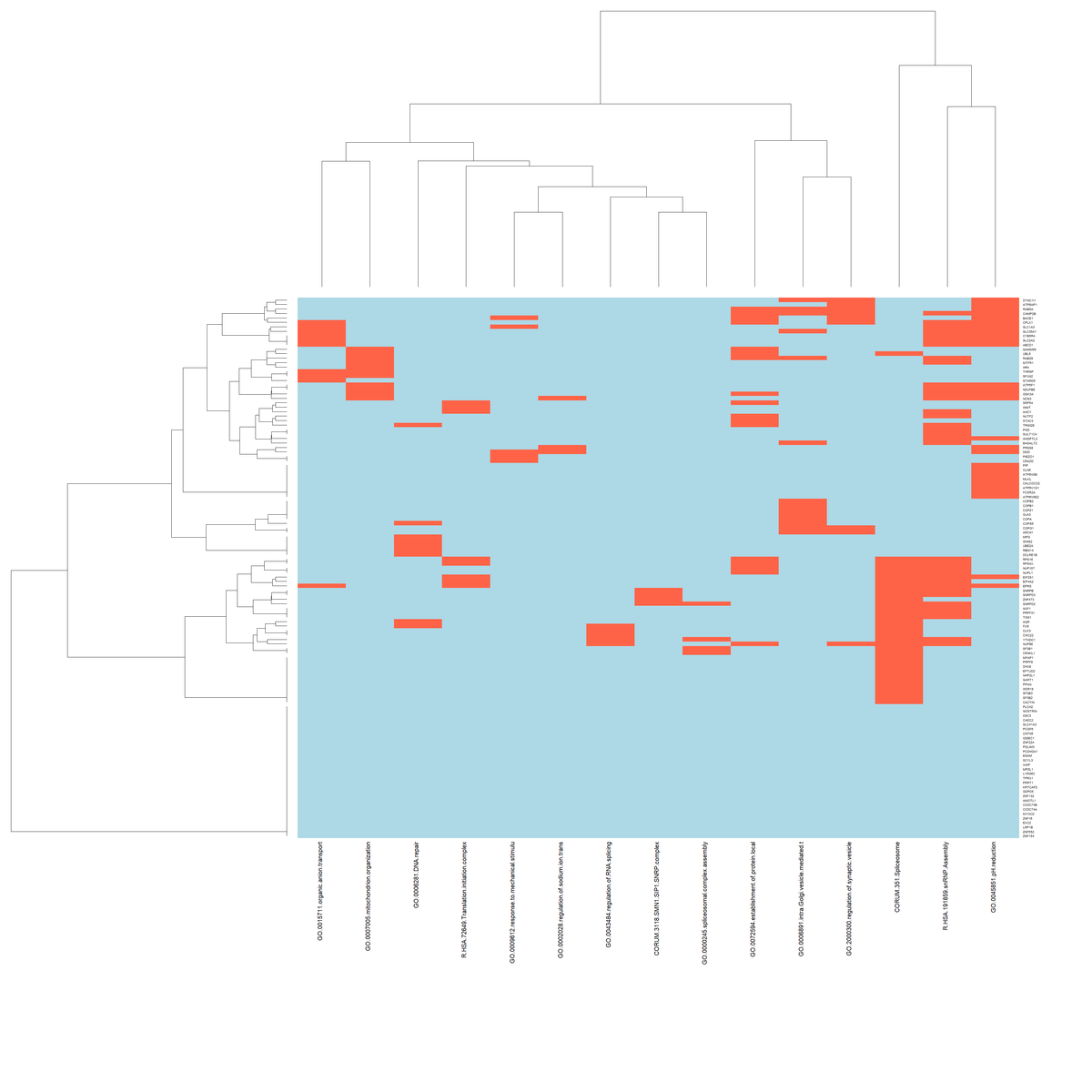
遺伝子のシグナル情報をもとに,同じ機能を持つ遺伝子をクラスタリングをすることができています.
今後はHCA解析などで作用メカ解析をする際にこのようなDBを活用していこうと考えています.
