こんにちは,@PKです.
今回はKNIMEで単回帰分析を行うWorkflowについて紹介します.
散布図・回帰直線の作成,回帰分析結果を出力し,どのノードを使うのが良いかを考えてみます.
KNIME Worlkflowの概要
今回紹介するWorkflowは以下の通りです.
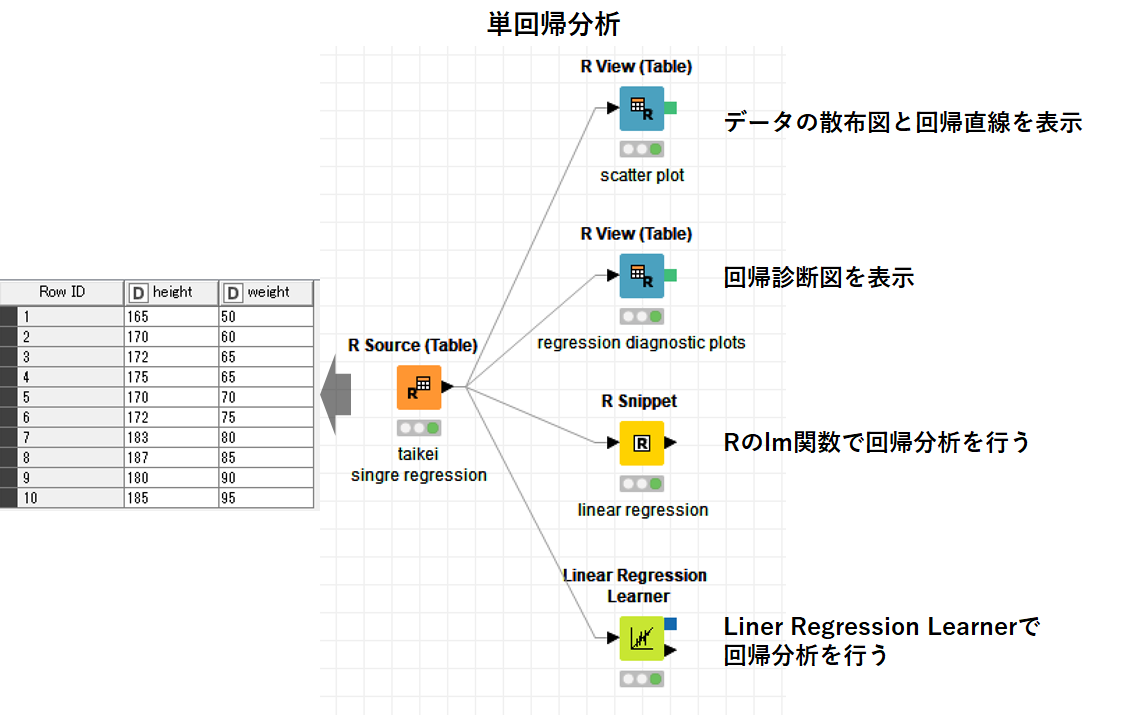
上図のデータを使って,散布図や回帰直線の作成,回帰分析結果の出力を行いたいと思います.
サンプルデータやRの解析については以下を参考にしております.
フリーソフトによるデータ解析・マイニング 第14回 Rと回帰分析
このWorkflowでは,単回帰分析について,データの可視化を「R view」で,それぞれの分析を「R snippet」と「Liner Regression Learner」で行います.
R View (Table) — NodePit
R Snippet — NodePit
Linear Regression Learner — NodePit
*このWorkflowでは統計解析向けのR言語を使用するノードを使います.
KNIMEでRを使用する際の基本的な部分は以下を参照ください.
【KNIME】Tukeyの多重検定を「R node」で処理・可視化する - t_kahi’s blog
「R view」で散布図と回帰直線の作成
まずは「R Source」でデータを作成します.
#R Sorce taikei = matrix(0,10,2) taikei[,1]<-c(165,170,172,175,170,172,183,187,180,185) taikei[,2]<-c(50,60,65,65,70,75,80,85,90, 95) colnames(taikei)<- c("height","weight") taikei.F<- data.frame(taikei) knime.out <- taikei.F
このデータは体重と身長の関係を表しています.
このデータの関係性を見るために,散布図と回帰直線を「R View」で表示します.
#R View library(ggplot2) library(gridExtra) #ggplot2の図を複数表示するため taikei <- knime.in data.lm<- lm(weight~height,taikei) #lm関数で回帰分析 plot1 <- ggplot(taikei, aes(x = height, y = weight)) + geom_point() + geom_abline(slope = data.lm$coefficients[2], intercept = data.lm$coefficients[1]) #ggplot2で散布図と回帰直線をマニュアルで作成 plot2 <- ggplot(taikei, aes(x = height, y = weight)) + geom_point() + geom_smooth(method = "lm") #geom_smoothで回帰直線作成 grid.arrange(plot1, plot2, ncol = 2, nrow = 1)
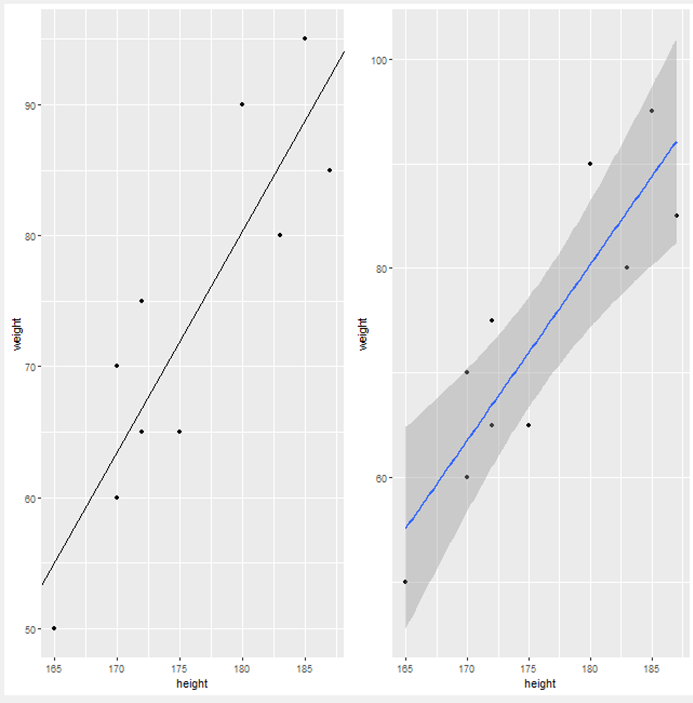 単回帰分析はRのlm関数を使用しました.
単回帰分析はRのlm関数を使用しました.
lm function | R Documentation
また,図の作成はggplot2を使用しています.
ggplot2による可視化入門
上図のような回帰直線はggplot2のgeom_abline(左図)やgeom_smooth(右図)で簡単に作成することができます.
単回帰分析の回帰診断図
続いて,回帰分析の残差を回帰診断図(Regression Diagnosis Plots)で表示します.
回帰診断図の作成については以下で紹介しております.
今回は結果のみを以下に示します.
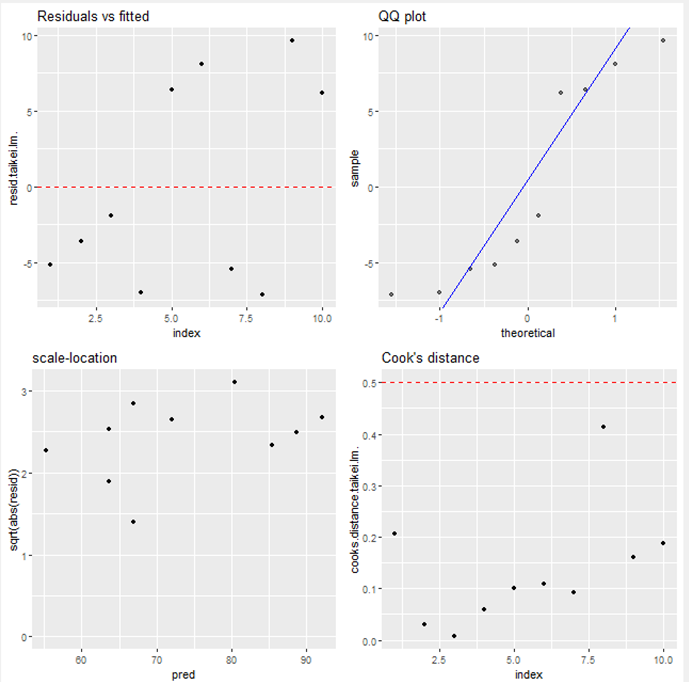
単回帰分析の結果を出力する
「Liner Regression Learner」
まずはKNIMEの線形回帰分析ノードである「Liner Regression Learner」で回帰分析を行います.
設定画面と結果を以下に示します.
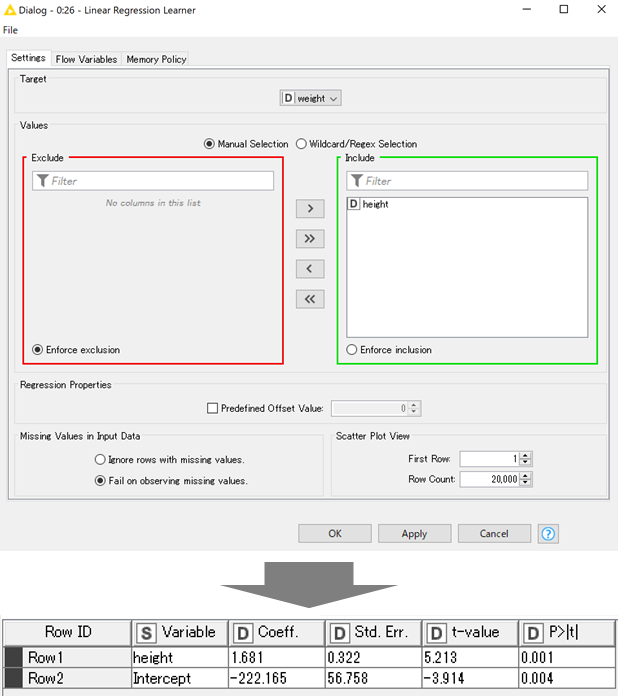
設定画面では,ターゲット列(被説明変数)を指定します.今回はWeightです.
Valuesの部分では説明変数のカラムを入れます.
また,このノード単体で散布図表示もできます.表示するデータはScatter Plot Viewsで設定できます.
「Liner Regression Learner」右クリック⇒Viewボタンをクリックで,下記の結果を見ることができます.
図はggplot2を使ってR viewsで表示させたほうがきれいですね…

「R snippet」
続いて,「R snippet」でRのlm関数を使って,単回帰分析の結果(回帰直線の切片,係数,t値,p値)を表示させます.
taikei <- data.frame(knime.in) taikei.lm<- lm(weight~height,data=taikei) summary(taikei.lm) taikei.lm$coefficients summary(taikei.lm)$coefficients knime.out <- summary(taikei.lm)$coefficients
出力結果は以下となります.
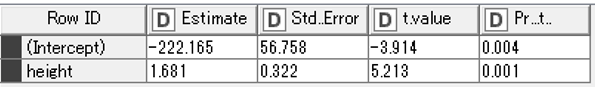
Rではsummary関数で結果を表示させますが,KNIMEでデータを出力するために,data.frame形式で値を取り出しています.
liner regressionのt値とp値を取得するのは以下の記事を参考にしました.
regression - Extracting t-stat p values from lm in R - Stack Overflow
「Liner Regression Learner」と「R snippet」の結果は(もちろん)一致しています.
まとめ
単回帰分析をKNIMEで行う場合ですが,
- 散布図,回帰直線の表示は「R views」のほうがきれい.
- 回帰分析結果の表示は「Liner Regression Learner」のほうが楽
というのが個人的な感想でした.
回帰診断図を書くノードは無いので,「R views」を使いましょう.