こんにちは,@PKです.
前回,CellProfilerの画像解析結果を,KNIMEで読み込み,Z-scoreの算出⇒可視化,というワークフローを紹介しました.
その際に,複数の特徴量をheatmapを使って可視化する処理について紹介しましたが,この特徴量は各細胞小器官の蛍光強度やサイズ,テクスチャなど多くの種類の情報を含んでおり,その数は約600個あります.
特徴量を複数取得することができるのは,他の評価系と比べてHigh Content Analysisの利点の一つです.
一方で,情報が重複している特徴量がある場合はどのように処理すれば良いのか,という課題がでてきます.
この課題の解決法として,今回は「Parameter Mutual Information」ノードについて紹介していきます.
イントロダクション
CellProfilerで取得できるオブジェクト(細胞内小器官など)の特徴量には以下のようなものがあります.
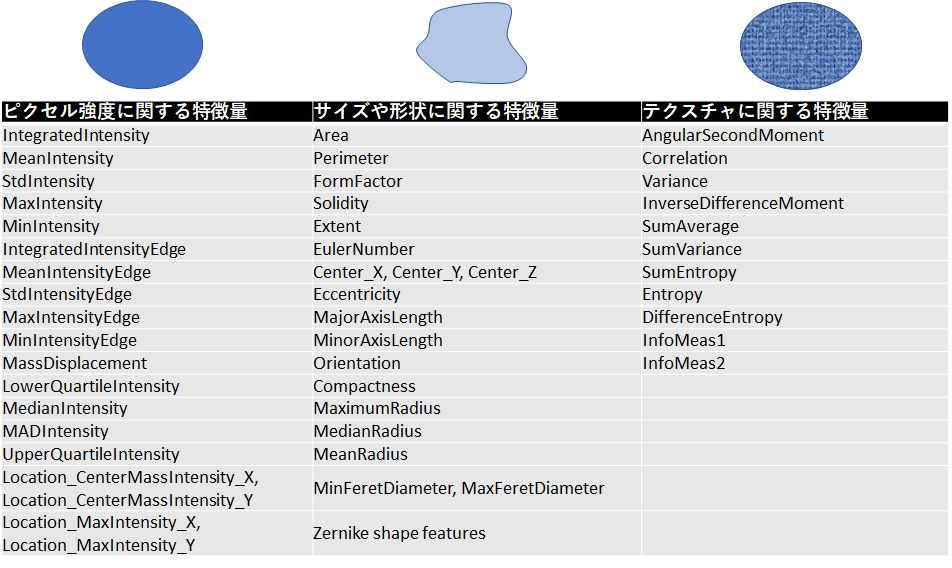 (*上記のパラメーターの取得については以下記事を参考にしてください)
(*上記のパラメーターの取得については以下記事を参考にしてください)
【CellProfiler】薬理活性化合物を処理した細胞染色画像から特徴量を抽出する② - t_kahi’s blog
核やミトコンドリアなど各オブジェクトごとに特徴量を取得するので,細胞全体の総特徴量は非常に多くなります.
前回の記事で紹介したheatmapをもう一度ここで見てみます. すると,いくつかの特徴量は類似した数値を示す(=重複している)ことが予想できます.

High contetnt screeningによって取得した複数の特徴量から,なるべく多くの情報をもれなくダブりなく取得したいです.
そのような場合に使うノードとして,「Parameter Mutual Information」がKNIMEに用意されていました.
「Parameter Mutual Information」ノード
概要とMutual Informationについて
「Parameter Mutual Information」はHCS tool⇒Pre-Processingの中にあります.
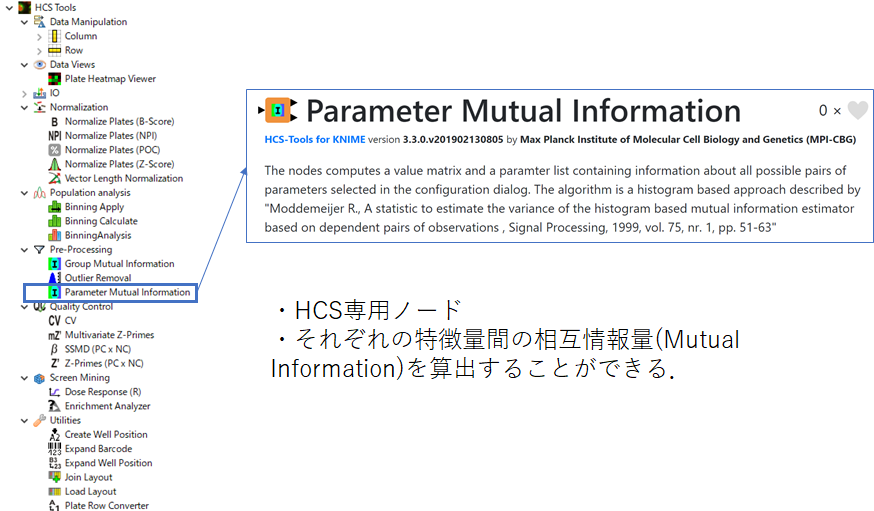
このノードはHCSのために作られたノードです.
各特徴量間の相互情報量(Mutual Information)を算出して,それぞれの特徴量間で,情報が重複しているかどうかを調べることができます.
Parameter Mutual Information — NodePit
数学に強くなかったので理解がおいついていませんが,このノードは以下の論文で示されているhistogram based approachを使用しており,各特徴量の情報量を計算してくれるようです.
そもそも,自分の場合は「相互情報量…?」となったのでいくつか日本語で説明している記事を読みました.
情報理論の基礎~情報量の定義から相対エントロピー、相互情報量まで~ | Logics of Blue
あれっ、エントロピーってなんだっけ?となったので復習した - Qiita
「21世紀の相関係数」を超える | NHN テコラス Tech Blog | AWS、機械学習、IoTなどの技術ブログ
非線形もOKな相関係数:MIC | Logics of Blue
これらの記事と論文の触りを読んで自分なりに解釈すると, 以下のようになりました.
- 相互情報量 (mutual information) は、ある事象を観測して、もう一つの事象に対する「予測しにくさ」がどれだけ減少したかを示している.
- 特徴量間で相互情報量が多ければ,片方の特徴量で,もう片方が予想できる,つまり同じような情報を持っていると判断することができる
- Mutual Informationはcorrelation coefficientの代わりに用いられる
- Mutual Informationはcorrelation coefficientと異なり,特徴量間に線形相関が無くても使える
勉強不足なので今後基礎的な部分を学んだら追記していきます. (もしおすすめの本などありましたら教えてください…)
Parameter Mutual Informationの設定
Parameter Mutual Informationノードの設定は以下のようになっています.
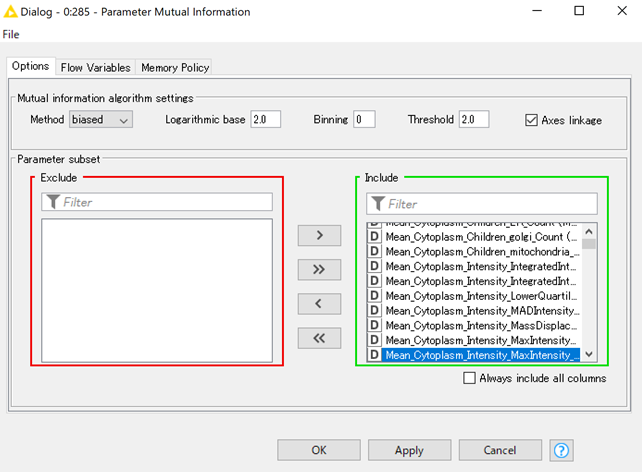
Mutual information algorithm setting:
Method : 相互情報量を測定するための手法
- unbiased (default)
- biased
- mmse (minimum mean square estimate)
*自分ノードを確認したところ,biasedがdefaultで設定されていました…
Logarithmic base : エントロピーを計算するための対数の低(デフォルトは2)
Binning : データを利参加するためのビニング分割の数(デフォルト⇒numberOfTableRows1/3)
数値が0の場合は,各パラメーターに対して相互情報量が計算される.
*こちらも自分のノードを確認したところ,defaultは0になっていました.Threshold:相互情報量がここで指定した値を超えると類似のパラメーターとみなされる閾値.(ビニングに応じて調整が必要)
Parameter subset:相互情報量を計算するカラムを選択する
ノードの説明と,自身のKNIMEで見たときのdefaultが若干異なっていたのがなぜなのかは不明でした…
Parameter Mutual Informationノードを使ってみる
前回紹介したZ-scoreを算出するワークフローで「Parameter Mutual Information」を使用してみます.
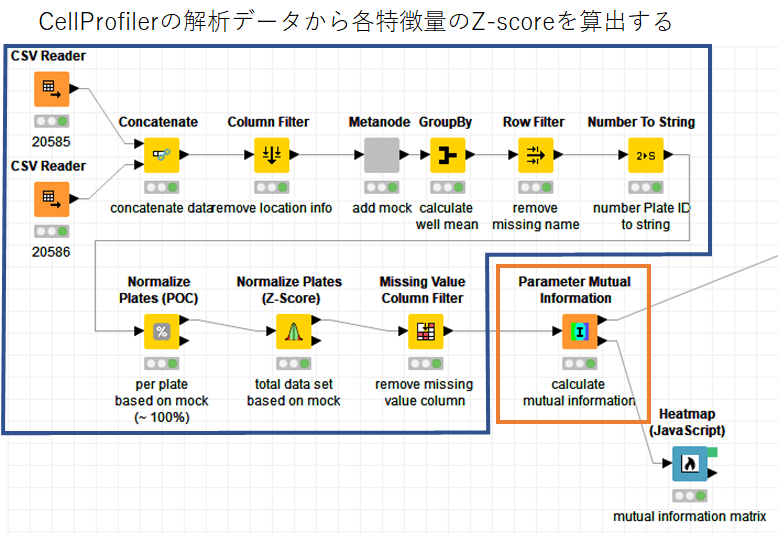
Mutual information algorithm settingはDefaultのままで行い,使用するパラメーターにはすべての特徴量を入れて解析を行いました.
このノードには2つの出力が用意されています.
まず上の出力ポートを表示すると以下のような相互情報量に関するリストが得られます.
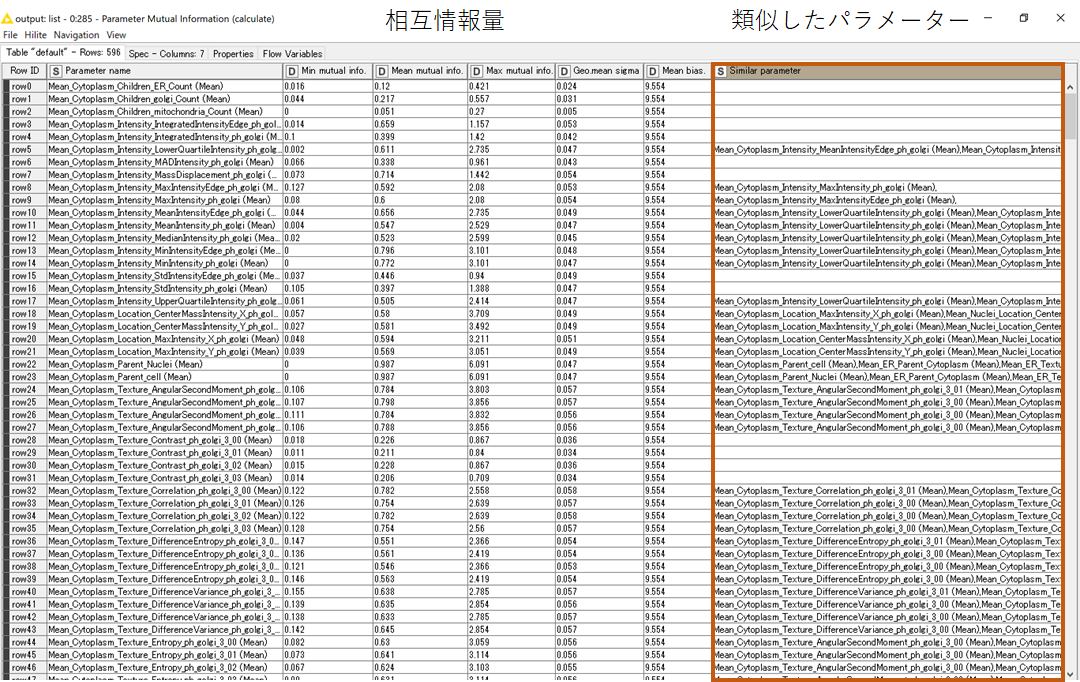
このリストでは,それぞれの特徴量の相互情報量を計算結果と,類似のパラメーターがあるかどうか示しています.
このデータを使って重複する特徴量を削減することができそうです.
また,下の出力ポートでは,パラメーターごとの相互情報量のマトリックスが得られます.
それぞれの特徴量ごとの相互情報量が示してあります.
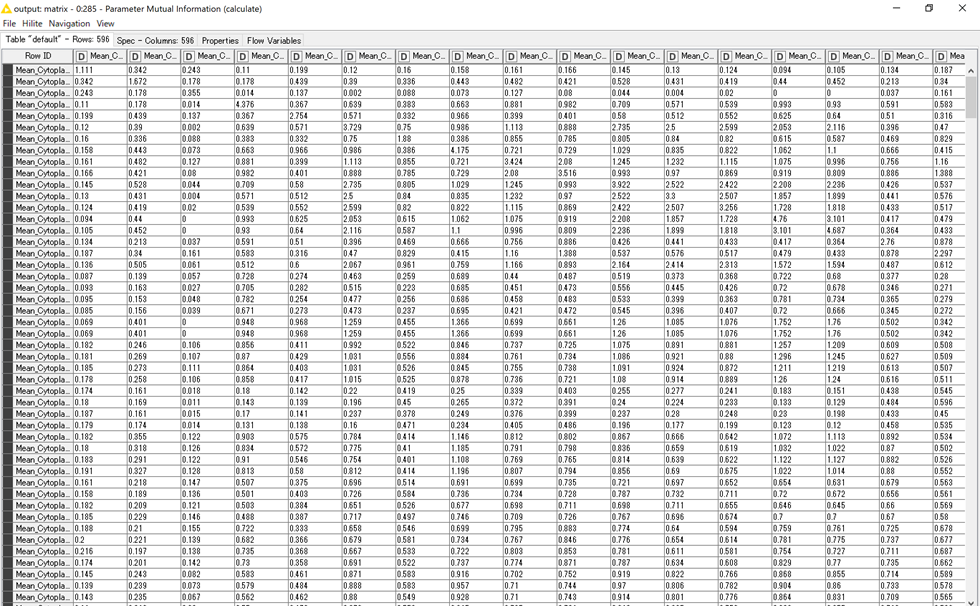
このままでは見づらいので,heatmapで表示した結果を示します.

各特徴量同士の相互情報量を可視化することができました.
そこで,実際にParameter Mutual Informationノードを使って重複した特徴量を除いてみました.
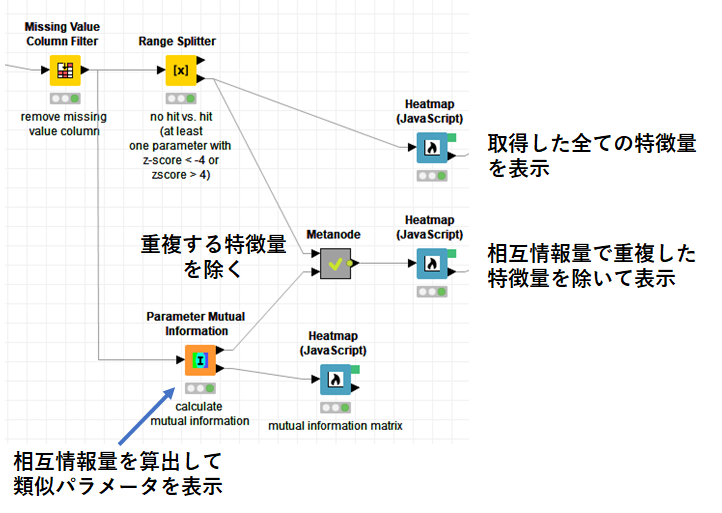
それぞれのheatmapを比較してみると,重複していた特徴量がまとまっています.

まとめ
HCS toolのParameter Mutual Informationノードを使うと,情報が重複した特徴量を見つけることができることを紹介しました.
HCSの解析に特化しており,非常に使いやすい反面,数学の知識がないと正しくデータを分析できているか不安になるので引き続き勉強していきます
おまけ:線形相関と比較する
KNIMEで線形相関見れないのかな…と思って調べてみると… 「Linear Correlation」というノードがありました.
せっかくなので「Parameter Mutual Information」と「Linear Correlation」で各特徴量の相互情報量や相関変数をheatmapで示しました.
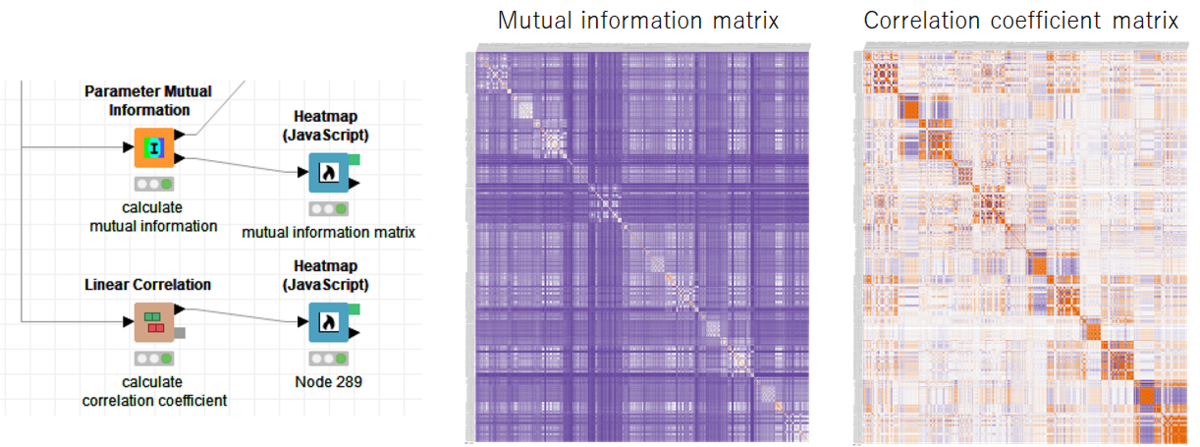
だいたい同じような分布なのですが,若干細かいパラメーターの値が異なっている気がしますね…この辺はしっかり勉強しないと良くわからないまま道具を使ってしまいそうで怖い…