こんにちは,@PKです.
以前に,CellProfilerを使って薬理活性化合物を処理した細胞染色画像の解析について紹介しました.
384well plate1枚分の画像を解析しただけですが,384well x 9field = 3456行のデータと数百の特徴量の列を含むデータなのでExcelで解析すると,煩雑で訳が分からなくなります.
汎用性もないので着々とExcelファイルが溜まってつらい思いをします.
そこで,登場するのがKNIMEです!
ここではCellProfilerの画像解析データからZ-Scoreを算出し,その結果を可視化するという取り組みについて紹介します.
はじめに
まず,今回の全体のフローを以下にお示しします.
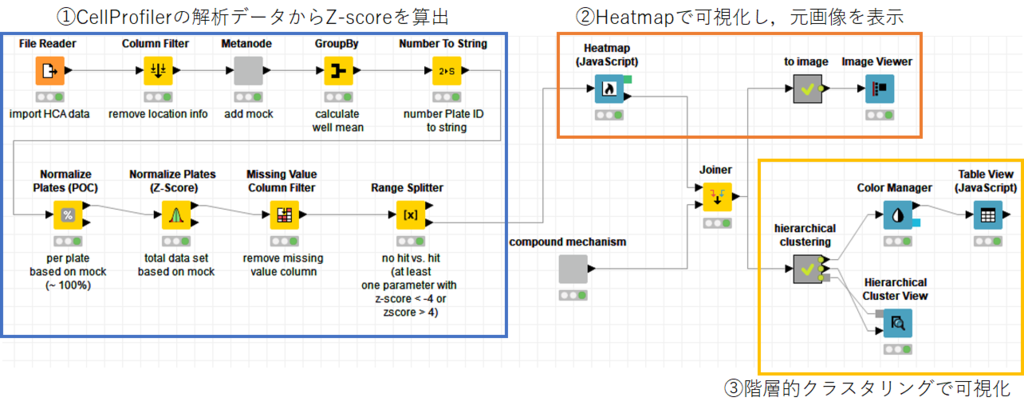
このワークフローは大きく3つの項目に分けることができます.
①CellProfilerの解析データから各特徴量のZ-scoreを算出する
②Heatmapでデータを可視化し,選択したデータの画像ファイルを表示する
③階層型クラスタリングで得られた結果を可視化する
この記事では①の各特徴量のZ-score算出について紹介しますが,何のためにこんな事しているのかを少し説明したいと思います.
この一連の解析のゴールは②や③のように得られたデータを分析し結果を解釈することです. ②については文字で説明し辛いので以下の2つの動画を参考にしてください.
このinteractive heatmapで薬理活性化合物ごとの特徴量(Z-score)を可視化しています. youtu.be
また,このノードはinteractive nodeなので,選んだ列のデータを次のフローに流すことができます. heatmapの結果は実際にどのような解析がされたのか?生データはどうなっているか?みたいなことを「Image viewer」で表示させることができます.
ここまで全てKNIME上で完結するの,かなりすごいと思いました.
このようなデータ分析を行うためには,CellProfiler解析データの前処理が必須になり,これが①の部分にあたります.
前置きが長くなりましたが,それでは①の処理について紹介していきます.
①ワークフローの紹介(前半)
それではワークフローを紹介していきます.
まずは以下の青線の部分についてです.
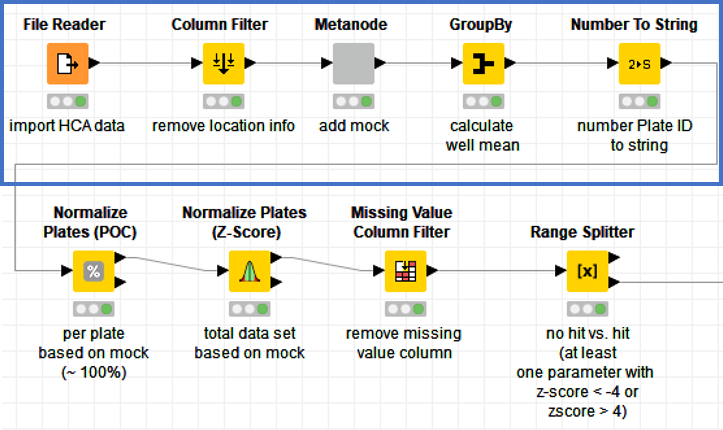
最初に「File Reader」で解析結果を読み込み,「Column Filter」で不必要なカラムを除去します.
今回使用するデータは,各視野毎の特徴量の平均値を出力したものです.
各細胞の位置情報(X,Y,Z)に関してはそもそも視野全体を平均化してしまっているので今回は解析対象から外します.「Column Filter」でそれらを除いて,約700の特徴量を残します.
続いてMetanodeの部分ですが,ここでは"mock",つまり無処理群の名前を変更しています.
簡単なノードの組み合わせなので,以下図に概要をまとめてしまいました.(もし細かいノードの中身が知りたい方がいたら連絡ください…)
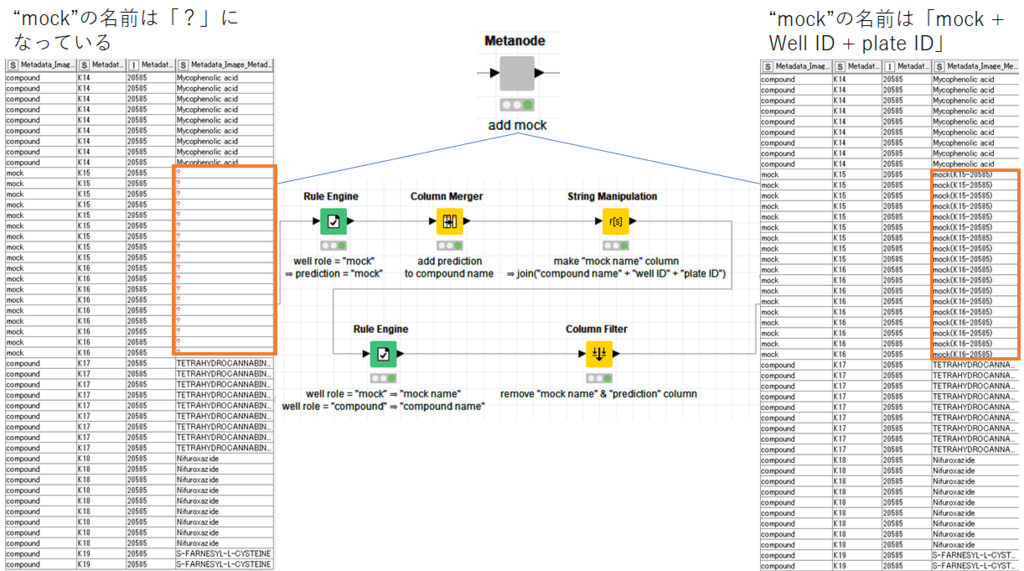
Metanodeの前のデータではassay well role = "mock"の化合物名の部分は「?」となっていますが,Metanodeの処理後は“mock”の名前は「mock + Well ID + plate ID」となっています.
これは自分が後で解析する際に各mock wellのばらつきがどの程度が知りたかったので,このような処理をしています.
単にmockはひとまとめにしても良いと思います.
続いて,「GroupBy」ノードで各視野毎のデータを(上図でも同じ化合物のデータが9視野分ある),各Plateの各Wellごとの平均値に変換します(以下図).
最後に「Number To String」ノードでPlate IDのカラムを数字列から文字列に変更します(次の正規化やZ-scoreの算出に必要)
①ワークフローの紹介(後半)
さて,後半のワークフローについて紹介していきます.
ここでは見慣れないノードがいくつか登場しています.
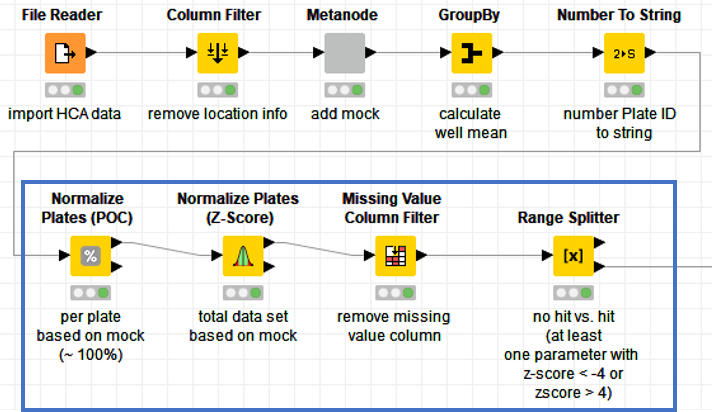
これらは「HCS-Tools」という拡張プラグインのノードです.
こんな感じでバイオ系のノードも充実しているのでKNIMEは楽しいですね.
High Content Screening Tools (trusted extension) | KNIME
HCS-Tools for KNIME [Plugin] — NodePit
「Normalize Plates(POC)」
まず,「Normalize Plates(POC)」というノードを使います.
Normalize Plates (POC) — NodePit
このノードでは,データ群を,あるサブセット(例:ネガティブコントロール)に対して正規化することができます.
今回は1枚のプレートの解析結果だけですが普通は複数のプレートのデータをまとめていくので,プレート間のばらつきが気になります.
そこで,各プレート間で化合物処理群をmockで正規化していきます.
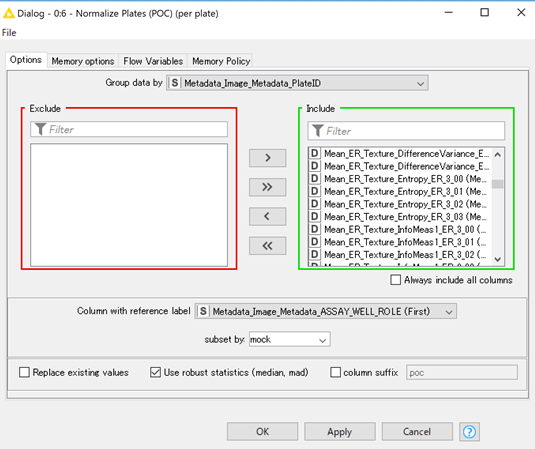
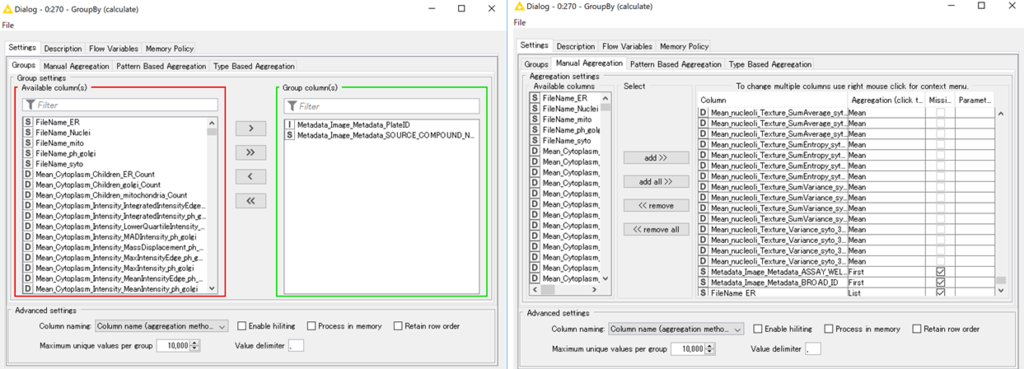
上図に示すように,
「Group data by」の部分に各プレートを定義する列を指定します.
「Column with reference label」の部分にどの列の情報を正規化に使うか指定し,「subset by」でmockを指定します.
Use robust statisticsにチェックを付けると中央値で正規化することができます.(x.poc = x / median(x[subset]) * 100)
「Normalize Plates(Z-score)」
続いて,「Normalize Plates(Z-score)」でZ-scoreを算出します.
Normalize Plates (Z-Score) — NodePit
Z-scoreの定義ですが,
分布の平均値からのずれを示す値.注目している標本値と分布の平均値の差を分布の標準偏差で割った値で定義される.Z-scoreの絶対値が大きければ大きい程,分布の平均値からのずれが大きいことを示している.
Z-score:バイオキーワード集|実験医学online:羊土社
このZ-scoreをスクリーニングに使用する目的ですが,以下のWikiの説明の通りで,Z-scoreを使うことで,ネガティブコントロール(mock)と比べて差がある化合物群を取得することができます.
「ヒット」の選択 HTSにおいて、効果の見込みが大きい化合物は「ヒット」といわれている。「ヒット」を選択するプロセスが、hit selection(「ヒット」の選択)といわれている。繰り返しがある場合と繰り返しがないスクリーニングでの、「ヒット」の選択のための分析方法は異なりる。繰り返しがないスクリーニングは通常は初めのスクリーニングで、繰り返しのあるスクリーニングは通常は確認のためのスクリーニングである。例えば、z-score method(Zスコア法)は反復のないスクリーニングに適し、t検定は反復のあるスクリーニングに適している。反復のないスクリーニングのための、SSMDの計算も、反復があるものとは異なる。
ハイスループットスクリーニング - Wikipedia
設定に関しては,「Normalize Plates(POC)」とほぼ同じです(以下図).
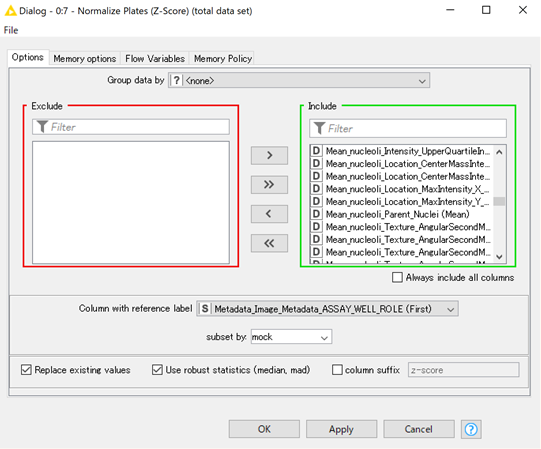
異なる点として,今度はプレート間ではなくて,全体をまとめて処理したいので,「Group data by」はnoneにしておきます.
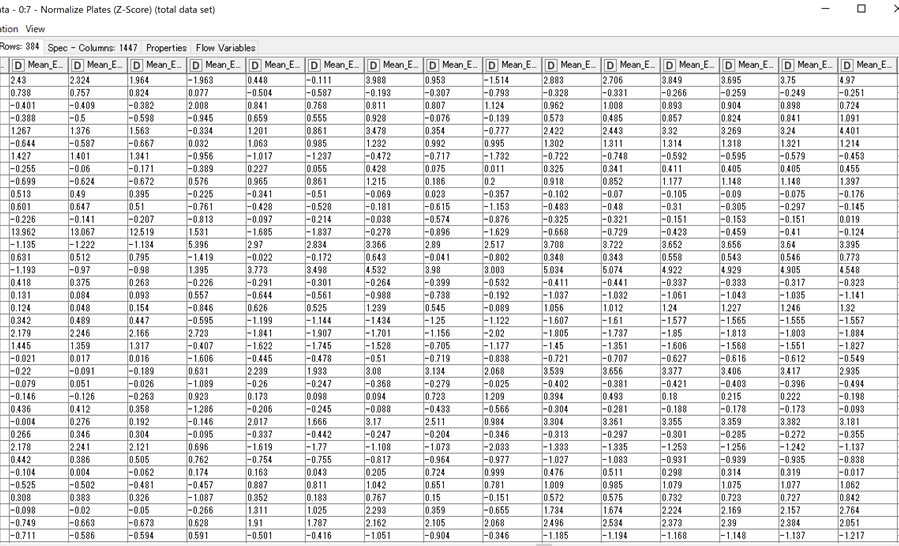
処理を流すと,以下のようにそれぞれの化合物ごとに各特徴量のZ-scoreが表示されているのがわかります.
「Missing Value Column Filter」⇒「Range Splitter」
続いて,「Missing Value Column Filter」ノードを使用します.
missing value(欠損値)が多い特徴量は解析に使用できないので,missing value threshold >90%と設定して,欠損値が9割以上含まれているカラムを除去します.
最後に「Range Splitter」ノードを使用して,各化合物処理群に対して,どれか一つでも良いので,各特徴量のZ-scoreが±4より大きい化合物をピックアップします.
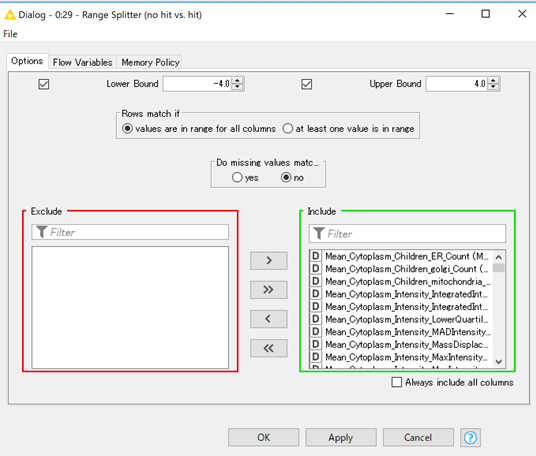
Z-scoreが±4より大きい化合物をとりたいのでLower Bound, Upper Boundに-4, +4をそれぞれ記載します.
続いて,「Rows match if」についてですが,以下の説明文の通りです.
"values are in range for all columns": Rows will be kept where all values of the chosen columns are within the given range. "at least one value is in range": Rows will be kept where at least one value of the chosen columns is within the given range.
今回は,一つでもZ-scoreの特徴量が外れている化合物群をピックアップしたいので,"values are in range for all columns"にチェックを入れます.
Z-scoreが±4以内の場合は,「Keep」へ,±4以上なら「Discard」へそれぞれの化合物処理群が分類されます.
勿論,今回欲しいのは「Discard」の部分に来る,mockと異なる特徴量を持つ化合物の情報です.
処理を流した後に,データを確認すると以下のように,215/384の列がZ-score±4以上に分類されました.
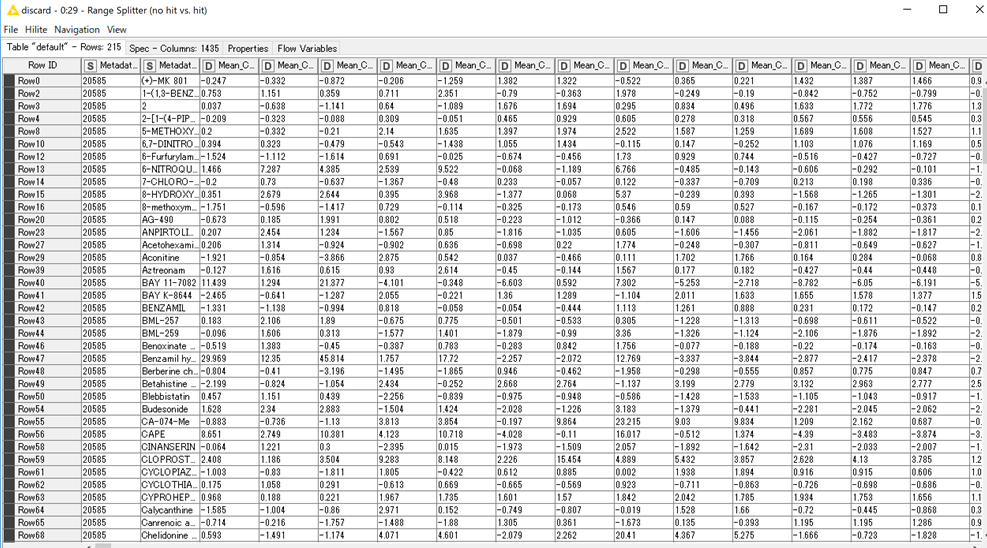
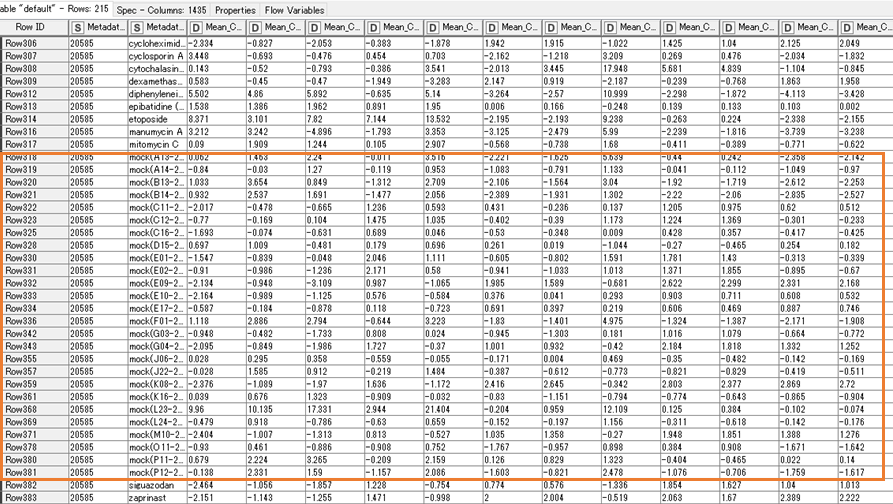
ちなみに…mockは384plateに64wellあったのですが,そのうち26がZ-score±4以上に分類されていました.意外とmockのばらつきが大きいのかな…とこのデータを見て感じました.
最後に
画像データを扱うのと比べると,この前処理はちょっと地味です…
ただ,この部分をしっかりやらないと結果の解析や分析はできないのでめちゃくちゃ重要な部分です.
データの解析手法は,ここで紹介したもの以外にもたくさんあります.
ここで紹介した解析手法が最適なのか,もっと良い解析手法はないか,を常に考えていこうと改めて思いました.
次回の記事ではいよいよ動画で紹介したheatmapや画像表示などの紹介をしたいと思います.