こんにちは,@PKです.
前回のブログでCellProfilerを使って細胞から様々な特徴量を抽出する取り組みを紹介しました.今回はその続きです. t-kahi.hatenablog.com
ミトコンドリアや小胞体,ゴルジ体などの細胞小器官のオブジェクト認識と,特徴量の抽出などを紹介していきます.
オブジェクトの紹介
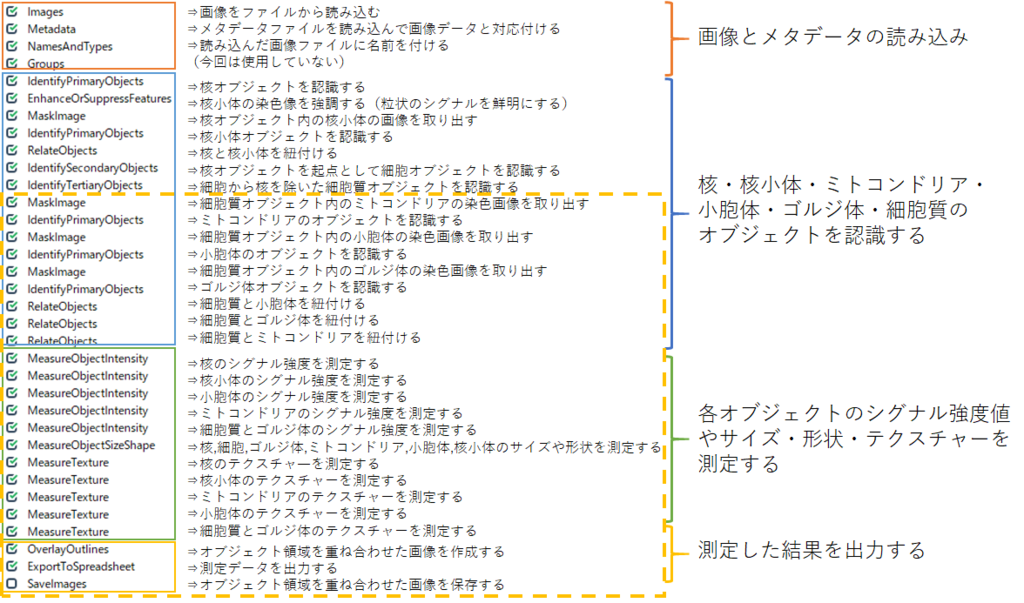
今回のパイプラインの流れをお示しします.黄色の点線囲いの部分が今日お話しする内容です.
ミトコンドリアと小胞体の認識
それではミトコンドリアと小胞体のオブジェクト領域の設定について紹介します.
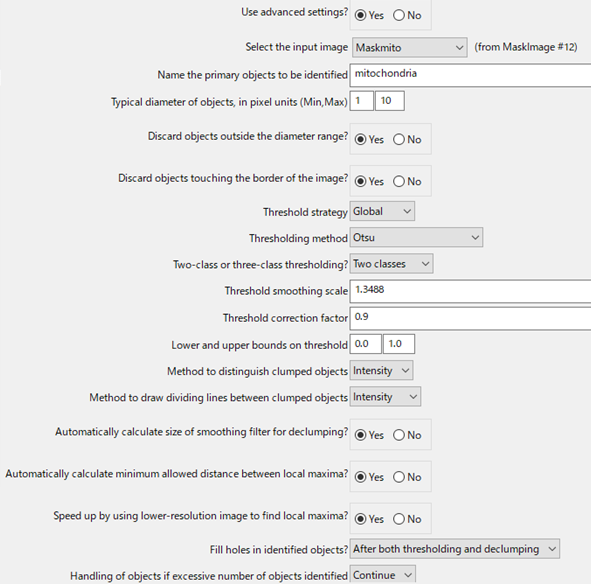
ミトコンドリアの「IdentifyPrimaryObjects」の設定は以下の通りです.
閾値の設定の部分は大津の二値化で行いました.
一番肝になるのは,Threshold correction factorという数値で,この部分を適当に変えながら自分が欲しい領域を設定していきます.今回は0.9という値を設定しました.
解析を行うと,ミトコンドリアの領域を抽出できていることがわかります.

小胞体に関しては,ミトコンドリアの設定とほとんど同じです.
閾値の設定だけは細かく見る必要があるので,いくつか試しました.
Threshold correction factorだけ0.8にして解析をすると,良い感じに領域が認識できました.

ゴルジ体の認識
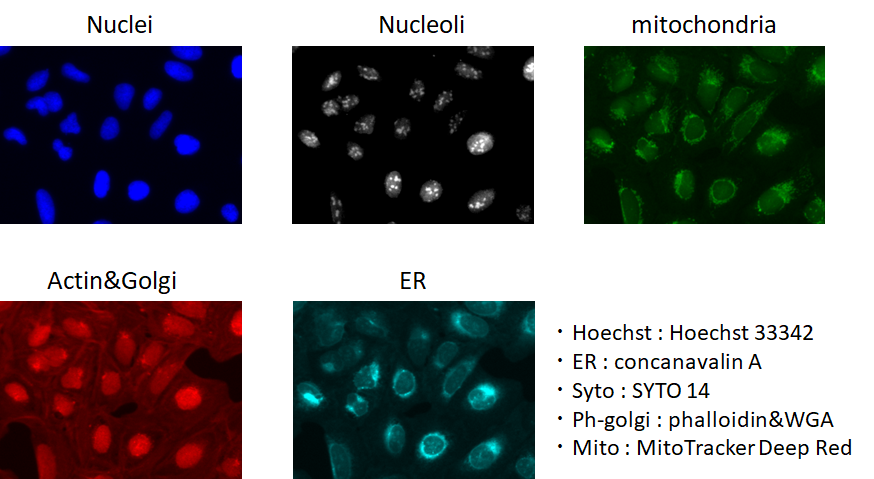
ゴルジ体の認識だけ,少しだけ注意が必要です.以下の図に示すようにゴルジ体のマーカーとアクチンは同じチャンネルで撮影されているので,そこから,ゴルジ体だけを分離する必要があります.
そこで,「IdentifyPrimaryObjects」を以下のように設定しました.
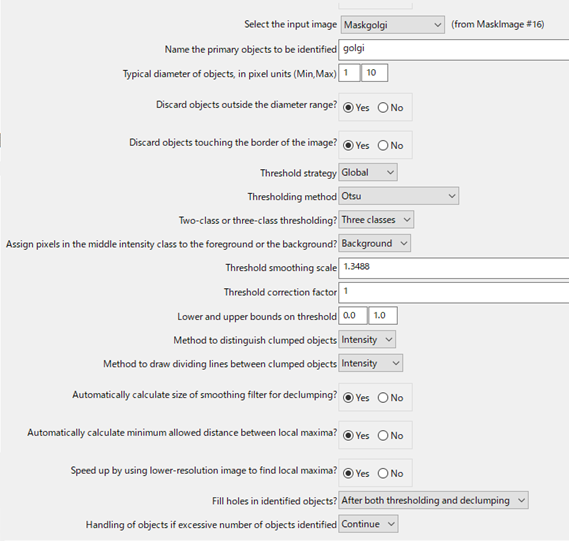 input imageの「Maskgolgi」の画像ですが,一つ前のMaskImageモジュールを使って,ゴルジ体とアクチン(細胞質)の染色画像から,細胞質の領域だけを抜き出しています.
input imageの「Maskgolgi」の画像ですが,一つ前のMaskImageモジュールを使って,ゴルジ体とアクチン(細胞質)の染色画像から,細胞質の領域だけを抜き出しています.
input imageの「MaskImage」は以下のように細胞外領域,細胞質,ゴルジ体のシグナルが観察されます.画像を見たときにゴルジ体は少しピクセル強度が若干高いことに着目しました.
閾値の設定で大津を選択した後に,Three classesに分け,middle intensity classをbackgroundへ設定することで,ゴルジ体の部分だけ抽出することができます.
実際に解析した結果が以下の通りです.一応,ゴルジ体の部分を取ってこれているかな..
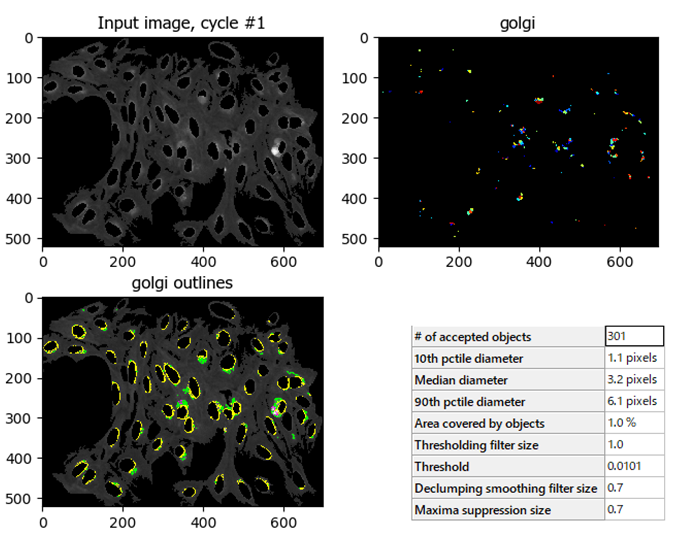 ちなみに,ここもThreshold correction factorがかなり重要でして,値を少し変えると結果もかなり変わることが下の図からわかると思います.
ちなみに,ここもThreshold correction factorがかなり重要でして,値を少し変えると結果もかなり変わることが下の図からわかると思います.

最後に「RelateObjects」で細胞質と核小器官を紐付けておきます.
特徴量について
ここでは,各オブジェクトのシグナル強度値やサイズ・形状・テクスチャーなどを測定します.
使用するのは,「MeasureObjectIntensity」「MeasureObjectSizeShape」「MeasureTexture」のモジュールです.
これらのモジュールによって取得できる特徴量について以下にまとめました.
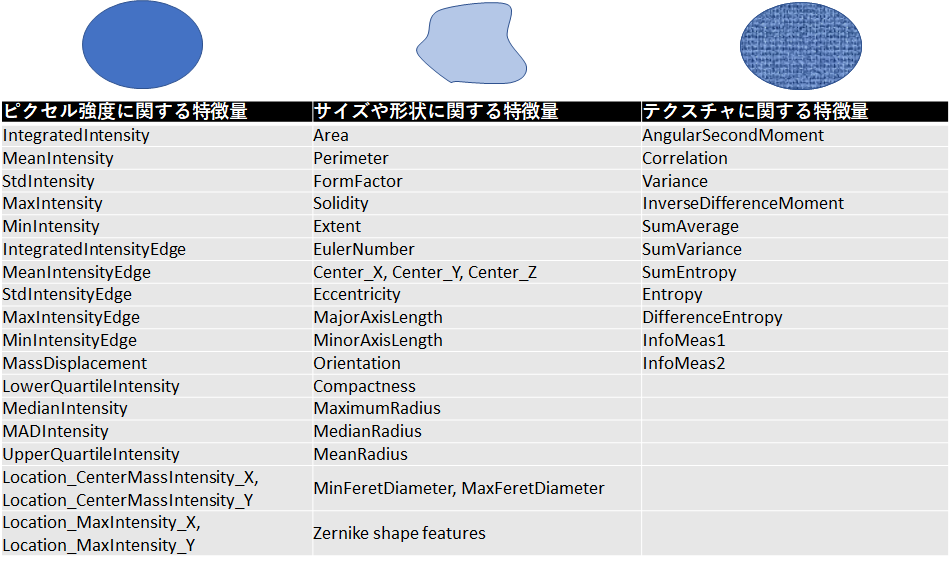
それぞれの説明は以下記事で触れているのでご興味があれば参考にしてください.
【CellProfiler】MeasureObjectIntensityで取得できる特徴量について - t_kahi’s blog 【CellProfiler】MeasureObjectSizeShapeで取得できる特徴量について - t_kahi’s blog 【CellProfiler】MeasureTextureモジュールについて - t_kahi’s blog
結果の出力について
CellProfilerで解析を行った際に,解析結果の数値データだけでなく,オブジェクトの重ね合わせ画像も取得することができます.
この重ね合わせ画像があると,数値データが変な値だった際に,生画像か,解析結果のどちらも確認をして判断することができます.
重ね合わせ画像を作成する「OverlayOutlines」のモジュールの設定画面を以下に示します.
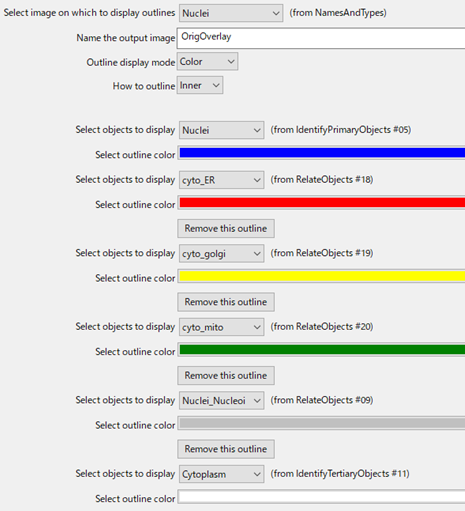
元となる画像と,重ね合わせたいオブジェクトを選んで解析を実行すると,以下のような画像を取得することができました.

最後にデータ出力ですが,「ExportToSpreadsheet」で数値データを出力し,「SaveImage」で画像データを出力します.
データを出力すると,以下のようにオブジェクトの重ね合わせ画像と,数値データを取得することができました!


最後に
「MeasureTexture」の解析がかなり時間がかかりました.
実際に384well x 5channel x 9field = 17280枚の画像解析をしたところ,自分のPCでは48時間以上もかかるという悲劇に…
これを機に良いPCを買いたいですね
ここで得られたデータをKNIMEで解析する取り組みを,次回以降のブログで紹介したいと思います.