こんばんは,@PKです.
前回の続きでExamplePipelineの処理を確認しながらCellProfilerの使い方に慣れていきます.
今回はPipeineの順番通りに各処理を確認していきますが,「Image」から「IdentifyPrimaryObjects」までを紹介したいと思います.
各パイプラインでどんな処理をしているか以下にに示します.
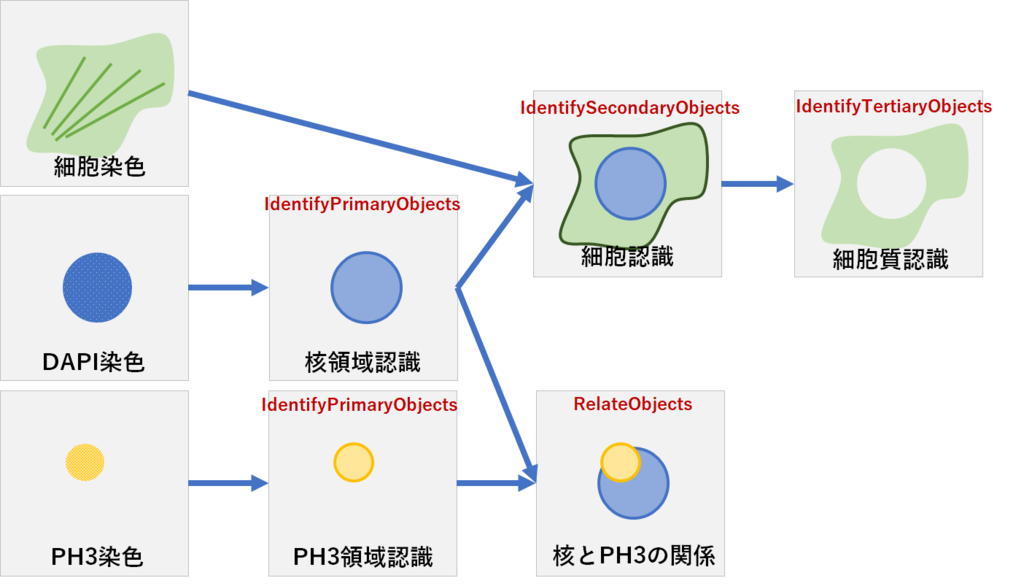

Image
まずは,画像の読み込みからです.
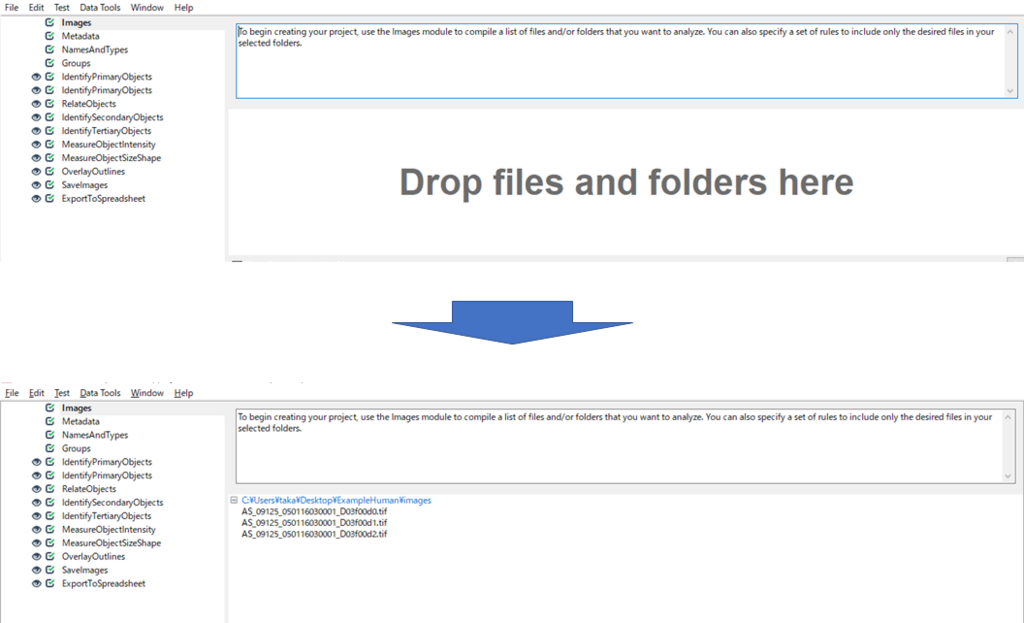
ここは非常に簡単で「image」モジュールを開くと「Drop files and folders here」という画面が出てきます.
ここに,読み込ませたい画像をフォルダごと入れるだけです.
また,画面をダブルクリックすると画像を選択できるので,そこから選んでもOKです.
ここでのポイントは,むしろどのような種類の画像を読み込ませるかだと思います.
解析できる画像は,デジタル画像(ある強度の値をもったピクセルが格子状に並んでいるもの)で,
などがあります.
また,画像ファイルの形式も重要なポイントです. CellProfilerは「Bio-Formats」というライブラリを使って様々フォーマットの画像ファイルを読むことができます.
Supported Formats — Bio-Formats 5.9.2 documentation
一般的に良く使う形式はTIF, PNG, JPG, JPEGなどだと思いますが,JPG/JPEGは情報が損失しているので,TIFやPNGなどの画像情報が失われないものが推奨されるようです.
NamesAndTypes
これは画像やチャンネルごとに名前を付けることができるモジュールです.
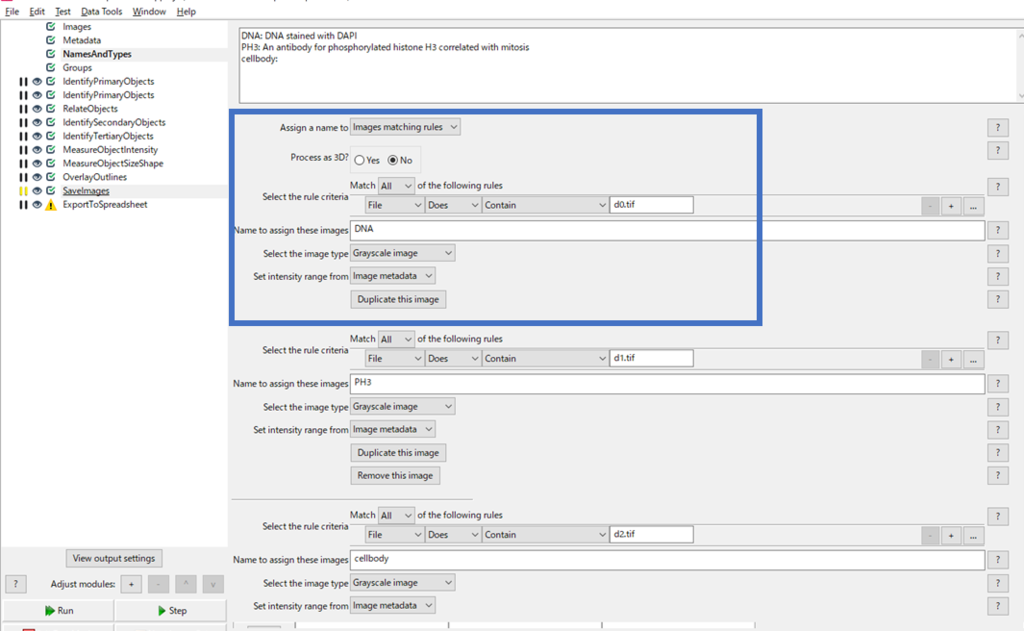
青枠の部分について簡単に説明します.
「Assign a name to 」で画像全てに名前を付けるのか,ルールに合致した画像だけ名前を付けるか選びます.
もし,共焦点顕微鏡などで撮ったZ-スタック画像を3次元解析したい場合は「Process as 3D」にチェックをいれます(今回はNo).
続いて,ファイルの名前ですが,「d0.tif」という名前がある画像にDNAと名前を付けています.
また,今回の画像はグレースケールなので,画像タイプを「Grayscale Image」に合わせておきます.
IdentifyPrimaryObjects(Nuclei)
ここでは,DNA染色の画像から,核オブジェクトの認識を行います.
まず,モジュールの中身を見ると「Used Advanced Setting」がNoになっています.このAdvanced Settingについては後ほど触れます.
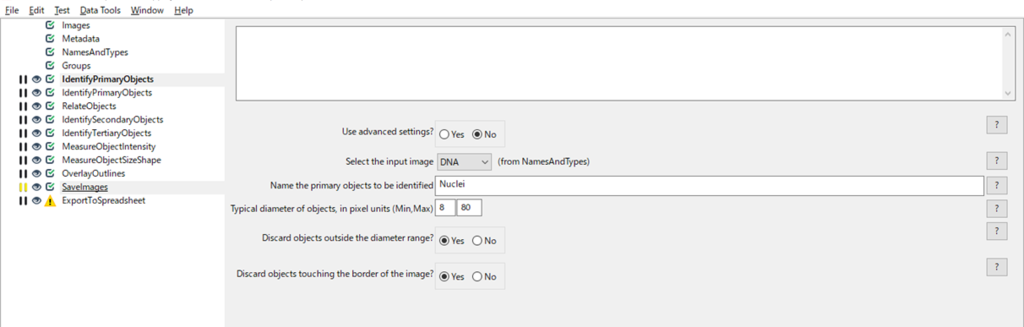
Basicでは簡単な設定だけを行っています. インプットしたい画像(DNA)を選び,オブジェクトの名前(Nuclei)を付け,その後オブジェクトの最小値・最大値の直径を選びます.
このオブジェクトのサイズは実際にIdentifyPrimaryObjectsを実行しながら値を調節していきましょう.
このモジュール実行ですが,以下の図の赤枠に2つのボタンがあります.
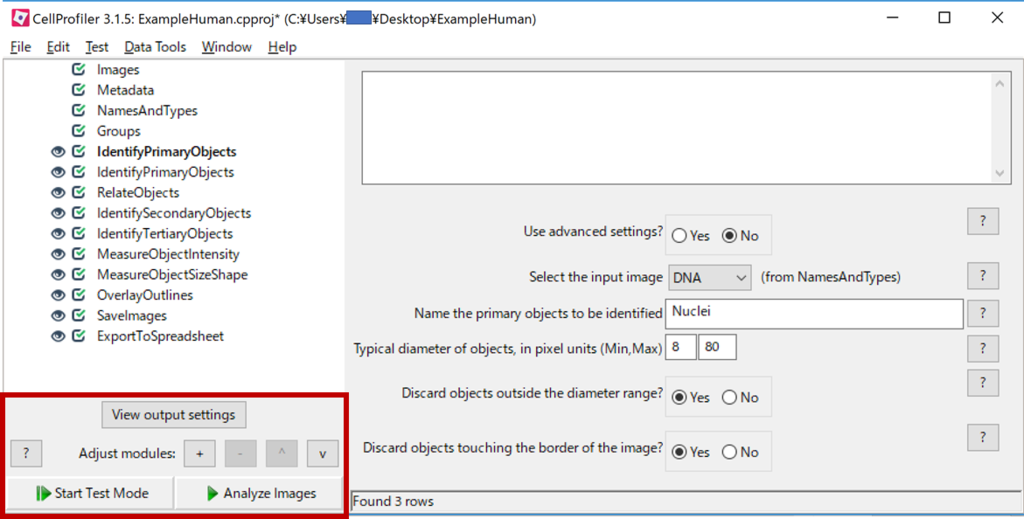
「▶Analyze Images」は全ての解析を実行します.
「▶Start Test Mode」は各モジュールごとに実行できるテストモードを開きます.
実際にボタンを押してみると,以下のように各モジュールの左側に一時停止ボタンが付いたのがわかると思います.
このボタンを押して黄色にした後に,上のモジュールを右クリックで「Run From module**」をクリックするとそこまでの解析を実行することができます.

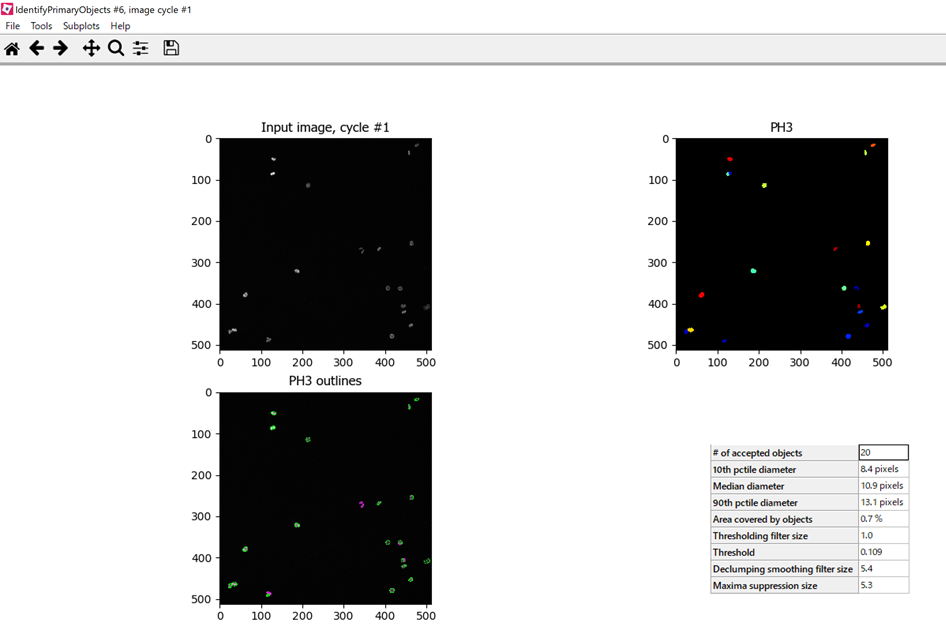
解析を実行すると解析結果が別ウィンドウで表示されます.
左上が生データ,右上が核オブジェクトの認識結果,左下が核オブジェクトの領域を示しています.
サンプルデータなのできれいに核領域が認識できているようですね…

上の虫眼鏡マークを押して,画像を拡大することができます. また,「Tools」⇒「Measure Length」を選ぶとオブジェクトの長さを図ることができます. 右下にLength : 14.9との表示が出ていて8-80の間に収まっていることがわかります.

「Discard objects outside the diameter range?」
⇒指定したレンジ外のオブジェクトを破棄するか
⇒基本的にはYesです.
「Discard objects touching the border of the image?」
⇒画像の端っこのオブジェクトは破棄するか
⇒ エリア値や蛍光強度は細胞全体が認識できている条件の値がしりたいのでYesにします.
IdentifyPrimaryObjects(PH3)
続いて,PH3のオブジェクトの認識です. こちらも基本的には核と同様なので割愛します.

画像解析を実行すると,以下のようにPH3のオブジェクトが認識できています.
ただ,拡大してみるとうまく認識されていない領域や,本来は1つなのに2つ認識されているような部分もあります. この辺は,解析者が画像データのどの領域を解析したいのか,に依存してきます.
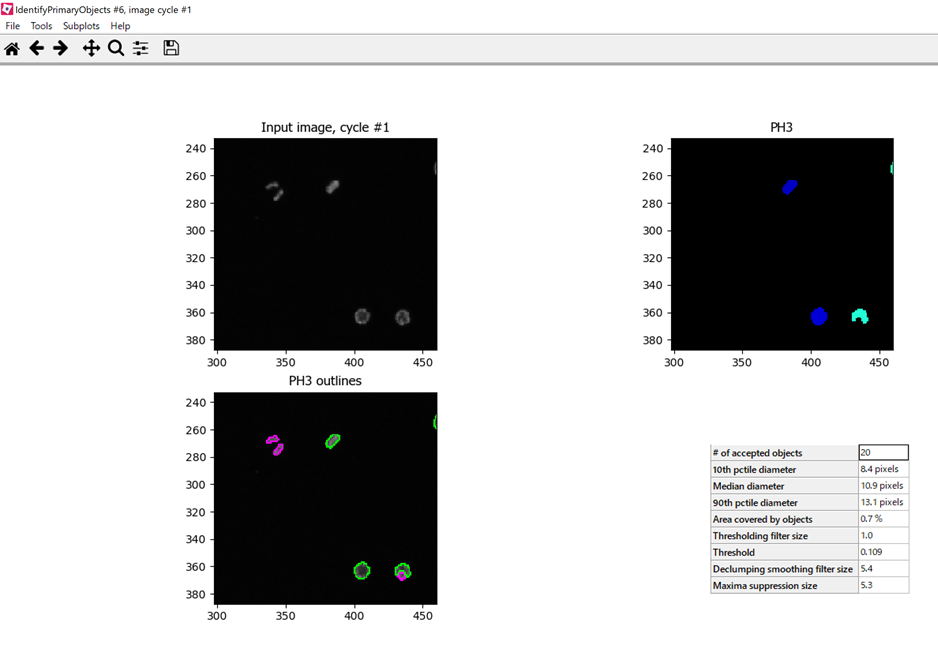
もう少し細かくオブジェクトの認識をしたい場合は,「Advanced Setting」を選択します.
こちらは少し内容が重たいので,次の記事で紹介したいとおもいます.